
COBAS HCV
Para visualizar el contenido completo de la IPPA (información para prescribir amplia), deberá iniciar sesión.
RESULTADOS:
El cobas® 5800 System y los cobas® 6800/8800 Systems determinan automáticamente la concentración de ARN del HCV en muestras y controles. La concentración de ARN del HCV se expresa en unidades internacionales por mililitro (UI/mL).
Control de calidad y validez de los resultados en el cobas® 5800 System:
• Con cada serie se procesan un control negativo ([–] C) y dos controles positivos, un control positivo bajo (HCV L[+]C) y un control positivo alto (HCV H[+]C) al menos cada 72 horas o con cada lote de kit nuevo. Los controles positivos y/o negativo pueden programarse con mayor frecuencia en función de los procedimientos de laboratorio y/o la reglamentación local.
• Compruebe los avisos y los resultados asociados tanto en el software cobas® 5800 como en el informe para garantizar la validez de la serie.
• La serie se considera válida siempre y cuando no haya avisos para ninguno de los tres controles, es decir, el control negativo y los dos controles positivos: HCV L(+)C, HCV H(+)C. El resultado del control negativo se muestra como (–) C, mientras que los controles positivos bajo y alto se presentan como HxV L(+)C y HxV H(+)C.
El software cobas® 5800 invalida automáticamente los resultados cuando los controles positivos y negativos fallan.
Nota: El cobas® 5800 System se suministra con la configuración estándar para el análisis de un conjunto de controles (positivo y negativo) con cada serie, pero se puede modificar por un programa menos frecuente de hasta cada 72 horas según los procedimientos de laboratorio y la reglamentación local. Póngase en contacto con su ingeniero técnico de Roche y/o con el represente del servicio técnico de Roche para obtener más información.
Resultados del control en el cobas® 5800 System: Los resultados de los controles se muestran en el software del cobas® 5800, en la aplicación de controles.
• Los controles se marcan como “Válido” en la columna “Resultados de control” cuando todas las dianas del control se han notificado como válidas. Los controles se marcan como “No válido” en la columna “Resultados de control” cuando todas o alguna de las dianas del control se han notificado como no válidas.
• Los controles marcados como “No válido” muestran un aviso en la columna “Aviso”. En la vista de detalles podrá encontrar más información sobre el motivo por el que el control se ha notificado como no válido, además de información sobre el aviso.
• Si uno de los controles positivos no es válido, repita el análisis de todos los controles positivos y de todas las muestras asociadas. Si el control negativo no es válido, repita el análisis de todos los controles y de todas las muestras asociadas.
Control de calidad y validez de los resultados en los cobas® 6800/8800 Systems:
• Con cada serie realizada en los cobas® 6800/8800 Systems se procesa un control negativo ([–] C) y dos controles positivos, un control positivo bajo (HCV L[+]C) y un control positivo alto (HCV H[+]C).
• Compruebe los avisos y los resultados asociados tanto en el software de los cobas® 6800/8800 Systems como en los informes para garantizar la validez de la serie.
• La serie se considera válida siempre y cuando no haya avisos para ninguno de los tres controles, es decir, el control negativo y los dos controles positivos: HCV L(+)C, HCV H(+)C. El resultado del control negativo se muestra como (–) C, mientras que los controles positivos bajo y alto se presentan como HxV L(+)C y HxV H(+)C.
El software cobas® 6800/8800 invalida automáticamente los resultados cuando los controles positivos y negativos fallan.
Avisos de controles en los cobas® 6800/8800 Systems:
Tabla 11. Avisos de controles para los controles negativo y positivo
|
Control negativo |
Aviso |
Resultado |
Interpretación |
|
(-) C |
Q02 (Serie de control errónea) |
No válido |
Un resultado inválido o el resultado del título calculado del control negativo no es negativo |
|
Control positivo |
Aviso |
Resultado |
Interpretación |
|
HxV L(+)C |
Q02 (Serie de control errónea) |
No válido |
Un resultado inválido o el resultado del título calculado para el control positivo bajo no está dentro del intervalo asignado |
|
HxV H(+)C |
Q02 (Serie de control errónea) |
No válido |
Un resultado inválido o el resultado del título calculado para el control positivo alto no está dentro del intervalo asignado |
Si la serie no es válida, repita las pruebas para toda la serie, incluyendo muestras y controles.
En el software de los cobas® 6800/8800 Systems, HxV L(+)C hace referencia al control positivo bajo cobas® HBV/HCV/HIV-1 y HxV H(+)C, al control positivo alto cobas® HBV/HCV/HIV-1.
Interpretación de los resultados: En las series de control válidas, compruebe cada muestra para detectar avisos en el software y/o los informes del cobas® 5800 System y de los cobas® 6800/8800 Systems. La interpretación de resultados se debe realizar del siguiente modo:
• Una serie válida puede incluir resultados de muestras tanto válidos como no válidos.
Tabla 12 Resultados de la diana para la interpretación de los resultados de la diana individuales
|
Resultados |
Interpretación |
|
Target Not Detected |
No se ha detectado ARN del HCV Los resultados se indican como “HCV no detectado” |
|
< Titer Min |
El título calculado está por debajo del límite inferior de cuantificación (LLoQ) del ensayo. Los resultados se indican como “HCV detectado, inferior a (título mínimo)” Título mínimo = 15 UI/mL (500 μL) Título mínimo = 40 UI/mL (200 μL) |
|
Titer |
El título calculado está comprendido en el intervalo lineal del ensayo: mayor o igual que el título mínimo, y menor o igual que el título máximo Los resultados se indican como “(Título) de HCV detectado” |
|
> Titer Maxa |
El título calculado está por encima del límite superior de cuantificación (ULoQ) del ensayo. Los resultados se indican como “HCV detectado, superior a (título máximo)” Título máximo = 1,00E+08 UI/mL (500 μL y 200 μL) |
a El resultado de muestra “> Titer Max” hace referencia a las muestras positivas para HCV detectadas con títulos superiores al límite de cuantificación superior (ULoQ). Si desea obtener resultados cuantitativos, diluya la muestra original con suero o plasma conservado en EDTA negativos al HCV (según el tipo de la muestra original) y repita la prueba. Multiplique el resultado comunicado por el factor de dilución.
Interpretación de los resultados en el cobas® 5800 System: Los resultados de las muestras se muestran en el software del cobas® 5800, en la aplicación de resultados.
En las series de control válidas, compruebe cada muestra para detectar avisos en el software cobas® 5800 System y/o en el informe. La interpretación de resultados se debe realizar del siguiente modo:
• Las muestras asociadas con las series de control válidas se muestran como “Válido” en la columna “Resultados de control” cuando todos los resultados de las dianas del control se han notificado como válidos. Las muestras asociadas con las series de control erróneas se muestran como “No válido” en la columna “Resultados de control” cuando todos los resultados de las dianas del control se han notificado como no válidos.
• Si los controles asociados de un resultado de muestra no son válidos, se añadirá un aviso específico al resultado de la muestra de la siguiente manera:
° Q05D: fallo en la validación de resultados debido a un control positivo no válido
° Q06D: fallo en la validación de resultados debido a un control negativo no válido
• Los valores en la columna “Resultados” para el resultado de la diana de la muestra individual deben interpretarse como se muestra en la Tabla 12 anterior.
• Si una o más dianas de la muestra están marcadas como “No válido”, el software cobas® 5800 muestra un aviso en la columna de avisos. En la vista de detalles podrá encontrar más información sobre el motivo por el que la(s) diana(s) de la muestra se ha notificado como no válidas, además de información sobre el aviso.
Interpretación de los resultados en los cobas® 6800/8800 Systems: En las series válidas, compruebe cada muestra para detectar avisos en el software cobas® 6800/8800 Systems y/o en el informe. La interpretación de resultados se debe realizar del siguiente modo:
• Las muestras presentan un “Yes” en la columna “Válida” si todos los resultados de las dianas solicitadas muestran resultados válidos. Las muestras que presentan un “No” en la columna “Válida” pueden requerir alguna interpretación o acción adicional.
• Los valores para el resultado de la diana de la muestra individual deben interpretarse como se muestra en la Tabla 12 anterior.
Limitaciones del procedimiento:
• La prueba COBAS® HCV se ha evaluado para ser utilizada únicamente con el cobas® HBV/HCV/HIV-1 Control Kit, el cobas® NHP Negative Control Kit, el cobas omni MGP Reagent, el cobas omni Lysis Reagent, el cobas omni Specimen Diluent y el cobas omni Wash Reagent en los cobas® 5800/6800/8800 Systems.
• La obtención de resultados fiables depende de que los procedimientos de recogida, almacenamiento y manipulación de muestras sean adecuados.
• Esta prueba se ha validado únicamente para su uso con muestras de suero o plasma conservado en EDTA. La realización de la prueba en otros tipos de muestras puede dar lugar a resultados inexactos.
• La cuantificación de ARN del HCV depende del número de partículas víricas presentes en las muestras y se puede ver afectada por los métodos de obtención de las mismas, factores propios del paciente (como la edad o la presencia de síntomas) y/o la fase de infección.
• Aunque es poco probable, las mutaciones en las regiones muy conservadas del genoma vírico cubiertas por la prueba COBAS® HCV pueden afectar la unión de cebadores y/o sondas, y causar una cuantificación a la baja del virus o incluso impedir su detección.
• Debido a las diferencias específicas entre tecnologías, se recomienda a los usuarios que realicen estudios de correlación en el laboratorio para determinar las diferencias tecnológicas antes de cambiar de una a otra. Los usuarios deberán adherirse a las políticas y los procedimientos específicos.
• La prueba COBAS® HCV no se ha concebido para el cribado de la presencia de HCV en sangre o hemoderivados.
OBTENCIÓN, TRANSPORTE Y ALMACENAMIENTO DE LAS MUESTRAS:
Nota: Manipule todas las muestras y los controles como si pudieran transmitir agentes infecciosos.
Almacene todas las muestras a las temperaturas especificadas.
La estabilidad de las muestras se ve afectada por las temperaturas elevadas.
Si se utilizan muestras congeladas en tubos secundarios, deje que se descongelen a temperatura ambiente (15-30 °C) por completo y, a continuación, agítelas unos instantes (entre 3 y 5 segundos) y centrifúguelas para que todo el volumen de la muestra se deposite en la parte inferior del tubo.
Muestras: La sangre debería recogerse en tubos de separación de suero SST™, en tubos para preparación de plasma BD Vacutainer® PPT™ para métodos de pruebas de diagnóstico molecular o en tubos estériles, y utilizar EDTA como anticoagulante. Siga las instrucciones del fabricante de los tubos de recogida de muestras.
• La sangre total recogida en tubos de separación de suero SST™, tubos para preparación de plasma BD Vacutainer® PPT™ para métodos de pruebas de diagnóstico molecular, o tubos estériles con EDTA como anticoagulante pueden almacenarse y/o transportarse durante un máximo de 24 horas a una temperatura comprendida entre 2 °C y 25 °C antes de la preparación del plasma/suero. La centrifugación debe realizarse conforme a las instrucciones del fabricante.
• Después de la separación, las muestras de plasma conservado en EDTA o suero pueden almacenarse en tubos secundarios durante un máximo de 6 días a una temperatura comprendida entre 2 y 8 °C, o un máximo de 12 semanas a ≤ -18 °C. Para un almacenamiento a más largo plazo se recomiendan temperaturas ≤ -60 °C.
• Las muestras de plasma/suero se mantienen estables hasta un máximo de cuatro ciclos de congelación/descongelación si se congelan a ≤ -18 °C.
• Si es posible, procure una cantidad de sangre total suficiente para permitir el uso del volumen de procesamiento preferido para suero o plasma conservado en EDTA de 500 μL (para un requisito de volumen de muestra mínimo total de 650 μL).
• Si las muestras se van a transportar, es recomendable empaquetarlas, y etiquetarlas de acuerdo con la reglamentación estatal y/o internacional relativa al transporte de muestras y agentes etiológicos.

Ilustración 1. Condiciones de almacenamiento de muestras
PRECAUCIONES Y REQUISITOS DE MANIPULACIÓN:
Advertencias y precauciones: Como sucede con cualquier procedimiento analítico, resulta esencial seguir las buenas prácticas de laboratorio recomendadas para obtener un rendimiento correcto del ensayo. Debido a la elevada sensibilidad de esta prueba, deben extremarse las precauciones para evitar cualquier tipo de contaminación de los reactivos y las mezclas de amplificación.
• Para diagnóstico in vitro exclusivamente.
• No se ha evaluado el uso de la prueba COBAS® HCV para el cribado de la presencia de HCV en sangre o productos sanguíneos.
• Todas las muestras de paciente deben tratarse como si fueran infecciosas, utilizando los procedimientos de laboratorio recomendados tal como se describe en la publicación Biosafety in Microbiological and Biomedical Laboratories y en el documento M29-A4 del CLSI.25,26 Solamente el personal competente en la manipulación de material biopeligroso, y en el uso de la prueba COBAS® HCV y los cobas® 5800/6800/8800 Systems deberían llevar a cabo este procedimiento.
• Todos los materiales de origen humano deben considerarse potencialmente infecciosos y manipularse teniendo en cuenta las precauciones generales. En caso de que se produzca un derrame, desinfecte de inmediato con una solución recién preparada de hipoclorito de sodio al 0,5% en agua destilada o desionizada (lejía doméstica diluida a 1:10), o siga los procedimientos apropiados del laboratorio.
• El cobas® HBV/HCV/HIV-1 Control Kit y el cobas® NHP Negative Control Kit contienen plasma procedente de sangre humana. Se ha analizado el material original mediante las pruebas para anticuerpos autorizadas y no se ha considerado reactivo para la presencia de anticuerpos del HCV, del HIV-1/2, HBsAg y anticuerpos anti-HBc. En el análisis del plasma humano normal con métodos de PCR no se ha detectado ARN de HIV-1 (grupos M y O), ARN de HIV-2, ARN de HCV ni ADN de HBV. Ningún método de prueba conocido puede garantizar totalmente que un producto derivado de la sangre humana no transmita agentes infecciosos.
• No congele la sangre entera ni las muestras almacenadas en tubos primarios.
• Utilice solo el material fungible suministrado o que se requiera expresamente para garantizar el óptimo rendimiento de la prueba.
• Puede solicitar Hojas de datos de seguridad (Safety Data Sheets [SDS]) al representante local de Roche.
• Siga al pie de la letra los procedimientos y las directrices que se suministran para garantizar la correcta realización de la prueba. Cualquier variación de dichos procedimientos y directrices podría afectar al rendimiento óptimo de la prueba.
• Podrían producirse resultados falsos positivos si no se evita la contaminación por arrastre de las muestras durante la manipulación y el procesamiento de las mismas.
• No utilice 200 μL de volumen de introducción de muestra si se espera que la carga viral sea < 100 UI/mL.
• Informe a la autoridad competente local de cualquier incidente grave que pueda tener lugar durante la realización del ensayo.
Manipulación de reactivos:
• Manipule todos los reactivos, controles y muestras de acuerdo con las prácticas de laboratorio recomendadas para evitar la contaminación por arrastre de las muestras o los controles.
• Antes de utilizarlos, revise cada casete de reactivo, diluyente, reactivo de lisis y reactivo de lavado para asegurarse de que no hay signos de fugas. No utilice el material si hay alguna evidencia de fuga.
• El cobas omni Lysis Reagent contiene tiocianato de guanidina, una sustancia química potencialmente peligrosa. Evite el contacto de reactivos con la piel, los ojos o las membranas mucosas. En caso de contacto, lave inmediatamente la zona afectada con abundante agua para evitar quemaduras.
• Los kits COBAS® HCV, el cobas omni MGP Reagent y el cobas omni Specimen Diluent contienen azida sódica como conservante. Evite el contacto de reactivos con la piel, los ojos o las membranas mucosas. En caso de contacto, lave inmediatamente la zona afectada con abundante agua para evitar quemaduras. Si se producen salpicaduras de reactivos, diluya las manchas con agua antes de secarlas con un paño.
• No permita que el cobas omni Lysis Reagent, que contiene tiocianato de guanidina, entre en contacto con la solución de hipoclorito de sodio (lejía). Tales mezclas pueden producir gases de alta toxicidad.
• Elimine todos los materiales que hayan estado en contacto con las muestras y los reactivos de acuerdo con la reglamentación nacional, estatal y local.
Buenas prácticas de laboratorio:
• No pipetee con la boca.
• No se debe comer, beber ni fumar en las áreas de trabajo.
• Utilice guantes, bata de laboratorio y protección ocular cuando manipule las muestras y los reactivos. Es necesario cambiarse los guantes entre la manipulación de las muestras y de los kits COBAS® HCV y los reactivos cobas omni para evitar la contaminación. Evite la contaminación de los guantes durante la manipulación de las muestras y de los controles.
• Lávese bien las manos después de manipular las muestras, los reactivos del kit y cuando se saque los guantes.
• Limpie y desinfecte minuciosamente todas las superficies de trabajo del laboratorio usando una solución recién preparada de hipoclorito de sodio al 0,5% en agua destilada o desionizada (lejía doméstica diluida a 1:10). A continuación, límpielas con un trapo impregnado en etanol al 70%.
• Si el derrame se produce sobre un instrumento cobas® 5800, siga las instrucciones descritas en la Asistencia al usuario y/o la Guía del usuario del cobas® 5800 System, para limpiar y descontaminar correctamente la superficie del instrumento.
• Si el derrame se produce sobre un instrumento cobas® 6800/8800, siga las instrucciones descritas en la Asistencia al usuario y/o la Guía del usuario de los cobas® 6800/8800 Systems, para limpiar y descontaminar correctamente la superficie de los instrumentos.
REACTIVOS Y MATERIALES:
Reactivos y controles de COBAS® HCV: Todos los reactivos y controles sin abrir deben almacenarse como se recomienda desde la Tabla 1 hasta la Tabla 4.
Tabla 1. COBAS® HCV
COBAS® HCV
Almacenar a 2-8 °C
Casete para 192 pruebas (P/N 09040765190)
|
Componentes del kit |
Composición del reactivo |
Cantidad por kit 192 pruebas |
|
Solución de proteinasa (PASE) |
Buffer tris, < 0,05% de EDTA, cloruro de calcio, acetato de calcio, 8% (p/v) de proteinasa EUH210: puede solicitarse la ficha de datos de seguridad EUH208: puede provocar una reacción alérgica Contiene: subtilisina, 9014-01-1 |
22,3 mL |
|
Estándar de cuantificación de ARN (RNA-QS) |
Buffer tris, < 0,05% de EDTA, < 0,001% de constructo de Armored RNA diferente del HCV que contiene regiones de secuencia específicas del cebador y la sonda (ARN no infeccioso encapsulado en bacteriófago MS2), < 0,1% de azida sódica |
21,2 mL |
|
Buffer de elución (EB) |
Buffer tris, 0,2% de metil-4-hidroxibenzoato |
21,2 mL |
|
Reactivo 1 de Master Mix (MMX-R1) |
Acetato de manganeso, hidróxido potásico, < 0,1% de azida sódica |
7,5 mL |
|
Reactivo 2 de Master Mix para HCV (HCV MMX-R2) |
Buffer tricina, acetato de potasio, 18% de sulfóxido de dimetilo, glicerol, < 0,1% de Tween 20, EDTA, < 0,12% de dATP, dCTP, dGTP y dUTP, < 0,01% de cebadores ascendente y descendente para HCV, < 0,01% de cebadores que van en un sentido y en sentido contrario para el estándar de cuantificación, < 0,01% de sondas oligonucleótidas marcadas con fluorescente específicas para HCV y el estándar de cuantificación del HCV, < 0,01% de aptámero oligonucleótido, < 0,01% de ADN polimerasa Z05D, < 0,1% de enzima AmpErase (uracil-N-glicosilasa) (microbiana), < 0,1% de azida sódica |
9,7 mL |
Tabla 2. cobas® HBV/HCV/HIV-1 Control Kit
cobas® HBV/HCV/HIV-1 Control Kit
Almacenar a 2-8 °C
Para uso en el sistema cobas® 5800 (P/N 09040773190)
Para uso en sistemas cobas® 6800/8800 (P/N 06997767190 y P/N 09040773190)
|
Componentes del kit |
Composición del reactivo |
Cantidad por kit |
Símbolo de seguridad y advertencia* |
|
Control positivo bajo para HBV/HCV/HIV-1 (HBV/HCV/HIV-1 L[+]C) |
< 0,001% de ARN (Armored) de HIV-1 grupo M encapsulado en proteína recubierta de bacteriófago MS2, < 0,001% de ADN (plásmido) sintético de HBV encapsulado en proteína recubierta de bacteriófago lambda, < 0,001% de ARN (Armored) sintético de HCV encapsulado en proteína recubierta de bacteriófago MS2, plasma humano normal, no reactivo según las pruebas autorizadas para anticuerpos frente al HCV, anticuerpos frente al HIV-1/2, HBsAg y anticuerpos anti-HBc; ARN de HIV-1, ARN de HIV-2, ARN de HCV y ADN de HBV no detectables mediante métodos de PCR 0,1% de conservante ProClin® 300** |
5,2 mL (8 × 0,65 mL) |
ADVERTENCIA H317: puede provocar una reacción alérgica en la piel H412: nocivo para los organismos acuáticos, con efectos nocivos duraderos P261: evitar respirar el polvo/el humo/el gas/la niebla/los vapores/el aerosol P273: evítese su liberación al medio ambiente P280: utilice guantes protectores P333 + P313: en caso de irritación o erupción cutánea: consultar a un médico P362 + P364: quitarse las prendas contaminadas y lavarlas antes de volver a usarlas P501: eliminar el contenido/el recipiente en una planta de eliminación de residuos autorizada 55965-84-9 masa de reacción de: 5-cloro-2-metil-4-isotiazolin-3-ona (n.º CE 247-500-7) y 2-metil-2H-isotiazol-3-ona (n.º CE 220-239-6) (3:1) |
|
Control positivo alto para HBV/HCV/HIV-1 (HBV/HCV/HIV-1 H[+]C) |
< 0,001% de ARN (Armored) sintético de título alto de HIV-1 grupo M encapsulado en proteína recubierta de bacteriófago MS2, < 0,001% de ADN (plásmido) sintético de HBV encapsulado en proteína recubierta de bacteriófago lambda, < 0,001% de ARN (Armored) sintético de HCV encapsulado en proteína recubierta de bacteriófago MS2, plasma humano normal, no reactivo según las pruebas autorizadas para anticuerpos frente al HCV, anticuerpos frente al HIV-1/2, HBsAg y anticuerpos anti-HBc; ARN de HIV-1, ARN de HIV-2, ARN de HCV y ADN de HBV no detectables mediante métodos de PCR 0,1% de conservante ProClin® 300** |
5,2 mL (8 × 0,65 mL) |
ADVERTENCIA H317: puede provocar una reacción alérgica en la piel P261: evitar respirar el polvo/el humo/el gas/la niebla/los vapores/el aerosol P272: las prendas de trabajo contaminadas no podrán sacarse del lugar de trabajo P280: utilice guantes protectores P333 + P313: en caso de irritación o erupción cutánea: consultar a un médico P362 + P364: quitarse las prendas contaminadas y lavarlas antes de volver a usarlas P501: Eliminar el contenido/el recipiente en una planta de eliminación de residuos autorizada 55965-84-9 masa de reacción de: 5-cloro-2-metil-4-isotiazolin-3-ona (n.º CE 247-500-7) y 2-metil-2H-isotiazol-3-ona (n.º CE 220-239-6) (3:1). |
* Las etiquetas de seguridad del producto se basan fundamentalmente en la regulación GHS de la UE.
** Sustancia peligrosa.
Tabla 3. cobas® NHP Negative Control Kit
cobas® NHP Negative Control Kit
Almacenar a 2-8 °C
Para uso en el sistema cobas® 5800 (P/N 09051554190)
Para uso en sistemas cobas® 6800/8800 (P/N 07002220190 y P/N 09051554190)
|
Componentes del kit |
Composición del reactivo |
Cantidad por kit |
Símbolo de seguridad y advertencia* |
|
Control negativo para plasma humano normal (NHP-NC) |
Plasma humano normal, no reactivo según pruebas autorizadas para anticuerpos del HCV, anticuerpos del HIV-1/2, HBsAg y anticuerpos anti-HBc; ARN de HIV-1, ARN de HIV-2, ARN de HCV y ADN de HBV no detectables mediante métodos de PCR < 0,1% de conservante ProClin® 300** |
16 mL (16 × 1 mL) |
ADVERTENCIA H317: puede provocar una reacción alérgica en la piel P261: evitar respirar el polvo/el humo/el gas/la niebla/los vapores/el aerosol P272: las prendas de trabajo contaminadas no podrán sacarse del lugar de trabajo P280: utilice guantes protectores P333 + P313: en caso de irritación o erupción cutánea: consultar a un médico P362 + P364: quitarse las prendas contaminadas y lavarlas antes de volver a usarlas P501: eliminar el contenido/el recipiente en una planta de eliminación de residuos autorizada 55965-84-9 masa de reacción de: 5-cloro-2-metil-4-isotiazolin-3-ona (n.º CE 247-500-7) y 2-metil-2H-isotiazol-3-ona (n.º CE 220-239-6) (3:1) |
* Las etiquetas de seguridad del producto se basan fundamentalmente en la regulación GHS de la UE.
** Sustancia peligrosa.
Reactivos cobas omni para la preparación de muestras:
Tabla 4. Reactivos cobas omni para la preparación de las muestras*
|
Reactivos |
Composición del reactivo |
Cantidad por kit |
Símbolo de seguridad y advertencia** |
|
cobas omni MGP Reagent (MGP) Almacenar a 2-8 °C (P/N: 06997546190) |
Partículas de vidrio magnéticas, buffer tris, 0,1% de metil-4 hidroxibenzoato, < 0,1% de azida sódica |
480 pruebas |
No aplicable |
|
cobas omni Specimen Diluent (SPEC DIL) Almacenar a 2-8 °C (P/N: 06997511190) |
Buffer tris, 0,1% de metil-4 hidroxibenzoato, < 0,1% de azida sódica |
4 × 875 mL |
No aplicable |
|
cobas omni Lysis Reagent (LYS) Almacenar a 2-8 °C (P/N: 06997538190) |
43% (p/p) de tiocianato de guanidina***, 5% (p/v) de polidocanol***, 2% (p/v) de ditiotreitol***, citrato de sodio dihidratado |
4 × 875 mL |
PELIGRO H302 + H332: nocivo en caso de ingestión o inhalación H314: provoca quemaduras graves en la piel y lesiones oculares graves H412: nocivo para los organismos acuáticos, con efectos nocivos duraderos EUH032: en contacto con ácidos libera gases muy tóxicos P261: evitar respirar el polvo/el humo/el gas/la niebla/los vapores/el aerosol P273: evítese su liberación al medio ambiente P280: llevar gafas/máscara de protección. P303 + P361 + P353: en caso de contacto con la piel (o el pelo): quitar inmediatamente todas las prendas contaminadas. Enjuagar la piel con agua P304 + P340 + P310: en caso de inhalación: transportar a la persona al aire libre y mantenerla en una posición que le facilite la respiración. Llamar inmediatamente a un centro de toxicología/médico P305 + P351 + P338 + P310: en caso de contacto con los ojos: aclarar cuidadosamente con agua durante varios minutos. Quitar las lentes de contacto, si lleva y resulta fácil. Seguir aclarando. Llamar inmediatamente a un centro de toxicología/médico 593-84-0 tiocianato de guanidina 9002-92-0 polidocanol 3483-12-3 (R*,R*)-1,4-dimercaptobutano-2,3-diol |
|
cobas omni Wash Reagent (WASH) Almacenar a 15-30 °C (P/N: 06997503190) |
Citrato de sodio dihidratado, 0,1% de metil-4-hidroxibenzoato |
4,2 L |
No aplicable |
* Estos reactivos no están incluidos en el kit de la prueba COBAS® HCV. Consulte el listado de material adicional necesario (Tabla 9).
** Las etiquetas de seguridad del producto se basan fundamentalmente en la regulación GHS de la UE.
*** Sustancia peligrosa.
Requisitos de almacenamiento de los reactivos: Los reactivos deben almacenarse y manipularse según las indicaciones de la Tabla 5, la Tabla 6 y la Tabla 7.
Cuando los reactivos no están cargados en el cobas® 5800 o los cobas® 6800/8800 Systems, almacénelos a la temperatura correspondiente especificada en la Tabla 5.
Tabla 5. Almacenamiento de reactivos (cuando el reactivo no está cargado en el sistema)
|
Reactivo |
Temperatura de almacenamiento |
|
COBAS® HCV |
2-8 °C |
|
cobas® HBV/HCV/HIV-1 Control Kit |
2-8 °C |
|
cobas® NHP Negative Control Kit |
2-8 °C |
|
cobas omni Lysis Reagent |
2-8 °C |
|
cobas omni MGP Reagent |
2-8 °C |
|
cobas omni Specimen Diluent |
2-8 °C |
|
cobas omni Wash Reagent |
15-30 °C |
Requisitos para la manipulación de reactivos en el cobas® 5800 System: Los reactivos cargados en el cobas® 5800 System se almacenan a la temperatura correspondiente adecuada y el sistema controla su fecha de caducidad. El sistema solamente permite utilizar los reactivos cuando se cumplen todas las condiciones indicadas en la Tabla 6. El sistema evita automáticamente el uso de reactivos caducados. La Tabla 6 ayuda al usuario a entender las condiciones de manipulación de los reactivos del cobas® 5800 System.
Tabla 6. Condiciones de caducidad de los reactivos del cobas® 5800 System
|
Reactivo |
Fecha de caducidad del kit |
Estabilidad del kit abierto |
Series en las que se puede utilizar el kit |
Periodo de estabilidad |
|
COBAS® HCV - 192 |
No caducado |
90 días desde el primer uso |
Máx. 40 series |
Máx. 36 días* |
|
cobas® HBV/HCV/HIV-1 Control Kit |
No caducado |
No aplicablea |
No aplicable |
Máx. 36 días* |
|
cobas® NHP Negative Control Kit |
No caducado |
No aplicablea |
No aplicable |
Máx. 36 días* |
|
cobas omni Lysis Reagent |
No caducado |
30 días desde la carga* |
No aplicable |
No aplicable |
|
cobas omni MGP Reagent |
No caducado |
30 días desde la carga* |
No aplicable |
No aplicable |
|
cobas omni Specimen Diluent |
No caducado |
30 días desde la carga* |
No aplicable |
No aplicable |
|
cobas omni Wash Reagent |
No caducado |
30 días desde la carga* |
No aplicable |
No aplicable |
a Reactivos de un solo uso.
* El tiempo se calcula desde la primera vez que se carga el reactivo en el cobas® 5800 System.
Requisitos para la manipulación de reactivos en los cobas® 6800/8800 Systems: Los reactivos cargados en los cobas® 6800/8800 Systems se almacenan a la temperatura correspondiente adecuada y el sistema controla su fecha de caducidad. Los cobas® 6800/8800 Systems solamente permiten utilizar los reactivos cuando se cumplen todas las condiciones indicadas en la Tabla 7. El sistema evita automáticamente el uso de reactivos caducados. La Tabla 7 ayuda al usuario a entender las condiciones de manipulación de los reactivos de los cobas® 6800/8800 Systems.
Tabla 7. Condiciones de caducidad de los reactivos de los cobas® 6800/8800 Systems
|
Reactivo |
Fecha de caducidad del kit |
Estabilidad del kit abierto |
Series en las que se puede utilizar el kit |
Periodo de estabilidad (horas acumuladas de carga fuera de la nevera) |
|
COBAS® HCV |
No caducado |
90 días desde el primer uso |
Máx. 40 series |
Máx. 40 horas |
|
cobas® HBV/HCV/HIV-1 Control Kit |
No caducado |
No aplicablea |
No aplicable |
Máx. 8 horas |
|
cobas® NHP Negative Control Kit |
No caducado |
No aplicablea |
No aplicable |
Máx. 10 horas |
|
cobas omni Lysis Reagent |
No caducado |
30 días desde la carga* |
No aplicable |
No aplicable |
|
cobas omni MGP Reagent |
No caducado |
30 días desde la carga* |
No aplicable |
No aplicable |
|
cobas omni Specimen Diluent |
No caducado |
30 días desde la carga* |
No aplicable |
No aplicable |
|
cobas omni Wash Reagent |
No caducado |
30 días desde la carga* |
No aplicable |
No aplicable |
a Reactivos de un solo uso.
* El tiempo se calcula desde la primera vez que se carga el reactivo en los cobas® 6800/8800 Systems.
Material adicional necesario para el cobas® 5800 System:
Tabla 8. Material y fungibles para el uso en el cobas® 5800 System
|
Material |
P/N |
|
cobas omni Processing Plate 24 |
08413975001 |
|
cobas omni Amplification Plate 24 |
08499853001 |
|
cobas omni Liquid Waste Plate 24 |
08413983001 |
|
Punta CORE TIPS con filtro, 1 mL |
04639642001 |
|
Punta CORE TIPS con filtro, 300 μL |
07345607001 |
|
cobas omni Liquid Waste Container |
07094388001 |
|
cobas omni Lysis Reagent |
06997538190 |
|
cobas omni MGP Reagent |
06997546190 |
|
cobas omni Specimen Diluent |
06997511190 |
|
cobas omni Wash Reagent |
06997503190 |
|
Bolsa para residuos sólidos o Bolsa para residuos sólidos con complemento |
07435967001 o 08030073001 |
Material adicional necesario para los cobas® 6800/8800 Systems:
Tabla 9. Materiales y fungibles para el uso en los cobas® 6800/8800 Systems
|
Material |
P/N |
|
cobas omni Processing Plate |
05534917001 |
|
cobas omni Amplification Plate |
05534941001 |
|
cobas omni Pipette Tips |
05534925001 |
|
cobas omni Liquid Waste Container |
07094388001 |
|
cobas omni Lysis Reagent |
06997538190 |
|
cobas omni MGP Reagent |
06997546190 |
|
cobas omni Specimen Diluent |
06997511190 |
|
cobas omni Wash Reagent |
06997503190 |
|
Bolsa para residuos sólidos y recipiente de residuos sólidos o Bolsa para residuos sólidos con complemento y kit del cajón |
07435967001 y 07094361001 o 08030073001 y 08387281001 |
Instrumentos y software necesarios: Es necesario instalar el software cobas® 5800 System y el paquete de análisis COBAS® HCV para el cobas® 5800 System en el instrumento cobas® 5800. El software Data Manager y el PC para el cobas® 5800 System se suministran con el sistema.
Es necesario instalar el software cobas® 6800/8800 Systems y el paquete de análisis COBAS® HCV para los cobas® 6800/8800 Systems en los instrumentos. El IG (instrument gateway) se suministra con el sistema.
Tabla 10. Instrumentos
|
Equipo |
P/N |
|
cobas® 5800 System |
08707464001 |
|
cobas® 6800 System (opción móvil) |
05524245001 y 06379672001 |
|
cobas® 6800 System (fijo) |
05524245001 y 06379664001 |
|
cobas® 8800 System |
05412722001 |
|
Módulo de suministro de muestras |
06301037001 |
Consulte la Asistencia al usuario y/o la Guía del usuario del cobas® 5800 System o los cobas® 6800/8800 Systems para obtener información adicional.
Nota: póngase en contacto con su representante local de Roche para obtener una lista de pedido detallada para racks de muestras, racks para puntas obstruidas y bandejas de racks compatibles con cada instrumento.
EVALUACIÓN CLÍNICA DEL RENDIMIENTO:
Variabilidad entre lotes y reproducibilidad: La variabilidad entre lotes y la reproducibilidad de la prueba COBAS® HCV se evaluaron con plasma conservado en EDTA en el cobas® 6800 System mediante un modelo mixto para estimar la variación total.
Los resultados se resumen a continuación desde la Tabla 27 hasta la Tabla 30.
Variabilidad entre lotes: Se realizaron pruebas de variabilidad entre lotes para los genotipos del 1 al 6 en un centro de pruebas con tres lotes de reactivo. Dos usuarios del centro analizaron cada lote durante 6 días. Se realizaron dos procesos por día.
La Tabla 27 se muestran los porcentajes atribuibles de la variación total, la SD de la precisión total y el CV en base log normal por genotipo y concentración esperada de ARN del HCV en log10 para el cobas® 6800 System.
Tabla 27. Porcentaje atribuible de la variación total, la desviación estándar de la precisión total y el CV (%) en base log normal de la concentración de ARN del HCV (log10 UI/mL) por genotipo y miembro positivo del panel en el cobas® 6800 System (entre lotes)
|
Genotipo |
Concentración de ARN de HCV |
N.º de pruebasb |
Porcentaje atribuible a la variación total (CV [%] log normal) |
Precisión total |
|||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
|
UI/mL esperada |
log10 en UI/mL esperada |
log10 UI/mL |
Lote |
Usuario |
Día |
Serie |
Intra-ensayo |
SDc |
CV (%) log normald |
||
|
1 |
30 |
1,477 |
1,482 |
68 |
0% (0,00) |
0% (0,00) |
0% (0,00) |
25% (22,14) |
75% (39,26) |
0,1899 |
45,91 |
|
100 |
2,000 |
1,890 |
72 |
8% (10,98) |
1% (3,68) |
0% (0,00) |
10% (12,12) |
81% (35,75) |
0,1672 |
39,97 |
|
|
5.000 |
3,699 |
3,457 |
72 |
0% (0,00) |
0% (0,00) |
0% (0,00) |
82% (32,85) |
18% (14,84) |
0,1531 |
36,38 |
|
|
50.000 |
4,699 |
4,443 |
72 |
3% (7,26) |
0% (0,00) |
0% (0,00) |
86% (37,29) |
11% (12,88) |
0,1693 |
40,51 |
|
|
500.000 |
5,699 |
5,552 |
72 |
0% (0,00) |
0% (0,00) |
0% (0,00) |
83% (33,86) |
17% (14,96) |
0,1570 |
37,36 |
|
|
5.000.000 |
6,699 |
6,453 |
71 |
47% (17,58) |
0% (0,00) |
0% (0,00) |
25% (12,71) |
28% (13,35) |
0,1100 |
25,74 |
|
|
50.000.000 |
7,699 |
7,103 |
72 |
54% (28,85) |
0% (0,00) |
0% (0,00) |
24% (19,14) |
22% (18,00) |
0,1670 |
39,92 |
|
|
2 |
30 |
1,477 |
1,611 |
72 |
5% (9,52) |
0% (0,00) |
8% (11,25) |
0% (0,00) |
87% (39,60) |
0,1776 |
42,67 |
|
100 |
2,000 |
2,125 |
72 |
0% (0,00) |
0% (0,00) |
0% (0,00) |
25% (12,12) |
75% (21,10) |
0,1047 |
24,47 |
|
|
5.000 |
3,699 |
3,714 |
72 |
9% (5,63) |
0% (0,00) |
0% (0,00) |
47% (12,66) |
44% (12,17) |
0,0798 |
18,53 |
|
|
50.000 |
4,699 |
4,743 |
72 |
0% (0,00) |
0% (0,00) |
0% (0,00) |
54% (16,10) |
46% (14,97) |
0,0949 |
22,12 |
|
|
500.000 |
5,699 |
5,806 |
72 |
7% (4,24) |
0% (0,00) |
0% (0,00) |
22% (7,39) |
71% (13,32) |
0,0684 |
15,85 |
|
|
5.000.000 |
6,699 |
6,187 |
72 |
41% (20,03) |
0% (0,00) |
0% (0,00) |
17% (12,73) |
42% (20,44) |
0,1348 |
31,80 |
|
|
50.000.000 |
7,699 |
7,080 |
72 |
40% (17,99) |
1% (2,73) |
0% (0,00) |
0% (0,00) |
59% (21,87) |
0,1223 |
28,73 |
|
|
3 |
30 |
1,477 |
1,474 |
72 |
0% (0,00) |
3% (8,35) |
0% (0,00) |
43% (32,35) |
54% (36,31) |
0,2084 |
50,89 |
|
100 |
2,000 |
1,946 |
72 |
13% (13,11) |
0% (0,00) |
0% (0,00) |
49% (25,49) |
38% (22,49) |
0,1562 |
37,16 |
|
|
5.000 |
3,699 |
3,636 |
72 |
14% (6,76) |
0% (0,00) |
0% (0,00) |
27% (9,30) |
59% (13,76) |
0,0776 |
18,01 |
|
|
50.000 |
4,699 |
4,597 |
72 |
0% (1,38) |
0% (0,00) |
0% (0,00) |
52% (14,95) |
47% (14,24) |
0,0894 |
20,80 |
|
|
500.000 |
5,699 |
5,504 |
72 |
0% (0,00) |
1% (1,62) |
0% (0,00) |
43% (13,51) |
57% (15,54) |
0,0893 |
20,77 |
|
|
5.000.000 |
6,699 |
6,451 |
72 |
28% (14,47) |
0% (0,00) |
3% (5,08) |
0% (0,00) |
69% (23,03) |
0,1189 |
27,91 |
|
|
50.000.000 |
7,699 |
7,149 |
71 |
21% (18,47) |
0% (0,00) |
8% (11,62) |
0% (0,00) |
71% (34,88) |
0,1747 |
41,90 |
|
|
4 |
30 |
1,477 |
1,358 |
69 |
7% (14,37) |
0% (0,00) |
1% (5,44) |
0% (0,00) |
91% (53,25) |
0,2269 |
56,03 |
|
100 |
2,000 |
1,827 |
72 |
10% (9,40) |
0% (0,00) |
1% (2,80) |
8% (8,35) |
81% (27,09) |
0,1283 |
30,21 |
|
|
5.000 |
3,699 |
3,416 |
72 |
20% (7,82) |
0% (0,00) |
0% (0,00) |
42% (11,23) |
38% (10,61) |
0,0750 |
17,40 |
|
|
50.000 |
4,699 |
4,405 |
72 |
22% (8,06) |
0% (0,00) |
0% (0,00) |
13% (6,30) |
65% (14,06) |
0,0752 |
17,46 |
|
|
500.000 |
5,699 |
5,069 |
71 |
5% (8,88) |
0% (0,00) |
24% (19,47) |
13% (14,23) |
57% (30,31) |
0,1699 |
40,66 |
|
|
5.000.000 |
6,699 |
6,070 |
72 |
27% (23,68) |
0% (0,00) |
12% (15,28) |
34% (26,55) |
27% (23,52) |
0,1940 |
47,00 |
|
|
50.000.000 |
7,699 |
6,930 |
72 |
37% (30,60) |
0% (0,00) |
22% (23,53) |
11% (16,70) |
30% (27,73) |
0,2149 |
52,68 |
|
|
5 |
30 |
1,477 |
1,575 |
72 |
5% (8,30) |
0% (0,00) |
0% (0,00) |
10% (11,53) |
85% (35,32) |
0,1611 |
38,42 |
|
100 |
2,000 |
2,049 |
72 |
9% (7,51) |
0% (0,00) |
0% (0,00) |
0% (0,00) |
91% (24,38) |
0,1093 |
25,57 |
|
|
5.000 |
3,699 |
3,606 |
72 |
4% (3,63) |
0% (0,00) |
0% (0,00) |
59% (14,11) |
38% (11,28) |
0,0797 |
18,51 |
|
|
50.000 |
4,699 |
4,616 |
72 |
20% (8,86) |
0% (0,00) |
0% (0,00) |
37% (12,19) |
43% (13,21) |
0,0867 |
20,17 |
|
|
500.000 |
5,699 |
5,678 |
72 |
7% (4,63) |
0% (0,00) |
0% (0,00) |
33% (10,36) |
60% (13,93) |
0,0777 |
18,04 |
|
|
5.000.000 |
6,699 |
6,505 |
71 |
54% (19,49) |
0% (0,00) |
19% (11,53) |
0% (0,00) |
27% (13,77) |
0,1143 |
26,79 |
|
|
50.000.000 |
7,699 |
7,592 |
72 |
35% (11,59) |
1% (2,25) |
12% (6,72) |
4% (3,94) |
47% (13,37) |
0,0842 |
19,58 |
|
|
6 |
30 |
1,477 |
1,494 |
70 |
0% (0,00) |
0% (0,00) |
0% (0,00) |
3% (7,34) |
97% (47,65) |
0,1990 |
48,33 |
|
100 |
2,000 |
1,940 |
72 |
9% (9,29) |
0% (0,00) |
0% (0,00) |
2% (4,14) |
90% (30,32) |
0,1361 |
32,13 |
|
|
5.000 |
3,699 |
3,417 |
72 |
0% (0,00) |
0% (0,00) |
0% (0,00) |
81% (37,28) |
19% (17,38) |
0,1737 |
41,64 |
|
|
50.000 |
4,699 |
4,541 |
72 |
0% (0,00) |
0% (0,00) |
0% (0,00) |
70% (26,40) |
30% (17,27) |
0,1351 |
31,88 |
|
|
500.000 |
5,699 |
5,611 |
72 |
0% (0,00) |
0% (0,00) |
0% (0,00) |
74% (22,82) |
26% (13,36) |
0,1136 |
26,62 |
|
|
5.000.000 |
6,699 |
6,414 |
72 |
49% (22,99) |
0% (0,00) |
9% (10,03) |
16% (12,88) |
26% (16,83) |
0,1413 |
33,42 |
|
|
50.000.000 |
7,699 |
7,529 |
71 |
48% (19,63) |
1% (2,67) |
2% (4,25) |
22% (13,15) |
28% (14,96) |
0,1225 |
28,78 |
|
Nota: La tabla únicamente incluye resultados con carga viral detectable.
a Calculadas mediante el procedimiento SAS MIXED.
b Número de pruebas válidas con carga viral detectable.
c Cálculo realizado utilizando la variabilidad total del procedimiento SAS MIXED.
d CV (%) log normal = sqrt(10^[SD^2 × ln(10)] - 1) × 100.
CV (%) = porcentaje del coeficiente de variación; HCV = virus de la hepatitis C; N.º = número; ARN = ácido ribonucleico; SD = desviación estándar; sqrt = raíz cuadrada.
En la Tabla 28 a continuación, el porcentaje de concordancia de negativos (PCN) del cobas® 6800 System con pruebas del miembro negativo del panel fue del 99,54%.
Tabla 28. Porcentaje de concordancia de negativos con el miembro negativo del panel en el cobas® 6800 System (entre lotes)
|
Concentración de ARN del HCV esperada |
N.º de pruebas |
Resultados positivos |
Resultados negativos |
Porcentaje de concordancia de negativosa |
IC del 95%b |
|
Negativa |
216 |
1 |
215 |
99,54 |
(97,45, 99,99) |
a Porcentaje de concordancia de negativos = (número de resultados negativos ÷ número total de pruebas válidas en el miembro negativo del panel) × 100.
b Calculado mediante el método de intervalo de confianza binomial exacto de Clopper-Pearson.
IC = intervalo de confianza; HCV = virus de la hepatitis C; N.º = número; ARN = ácido ribonucleico.
Reproducibilidad: Las pruebas de reproducibilidad se realizaron en tres centros para los genotipos del 1 al 3 utilizando un lote de reactivo. Dos operadores de cada centro realizaron las pruebas durante seis días. Se realizaron dos procesos por día.
En la Tabla 29 se muestran los porcentajes atribuibles de la variación total, la SD de la precisión total y el CV en base log normal por genotipo y concentración esperada de ARN del HCV en log10 en el cobas® 6800 System.
Tabla 29. Porcentaje atribuible de la variación total, la desviación estándar de la precisión total y el CV (%) en base log normal de la concentración de ARN del HCV (log10 UI/mL) por genotipo y miembro positivo del panel en el cobas® 6800 System (reproducibilidad)
|
Genotipo |
Concentración de ARN de HCV |
N.º de pruebasb |
Porcentaje atribuible a la variación total (CV (%) log normal) |
Precisión total |
|||||||
|
UI/mL esperada |
log10 en UI/mL esperada |
log10 UI/mL |
Centro |
Usuario |
Día |
Serie |
Intra-ensayo |
SDc |
CV (%) log normald |
||
|
1 |
30 |
1,477 |
1,373 |
68 |
1% (6,43) |
0% (0,00) |
0% (0,00) |
20% (25,63) |
78% (52,96) |
0,2437 |
60,84 |
|
100 |
2,000 |
1,866 |
72 |
4% (7,25) |
0% (0,00) |
0% (0,00) |
17% (15,81) |
79% (34,64) |
0,1644 |
39,24 |
|
|
5.000 |
3,699 |
3,466 |
72 |
0% (0,00) |
0% (0,00) |
0% (0,00) |
83% (29,77) |
17% (13,35) |
0,1391 |
32,87 |
|
|
50.000 |
4,699 |
4,444 |
72 |
7% (10,74) |
0% (0,00) |
0% (0,00) |
83% (37,40) |
9% (12,16) |
0,1721 |
41,24 |
|
|
500.000 |
5,699 |
5,579 |
72 |
4% (6,84) |
0% (0,00) |
0% (0,00) |
74% (30,53) |
22% (16,27) |
0,1504 |
35,70 |
|
|
5.000.000 |
6,699 |
6,439 |
72 |
52% (16,35) |
9% (6,91) |
0% (0,00) |
9% (6,74) |
30% (12,36) |
0,0979 |
22,84 |
|
|
50.000.000 |
7,699 |
7,091 |
72 |
76% (45,80) |
0% (0,00) |
0% (0,00) |
7% (12,87) |
17% (20,92) |
0,2170 |
53,25 |
|
|
2 |
30 |
1,477 |
1,631 |
72 |
10% (11,41) |
0% (0,00) |
0% (0,00) |
0% (0,00) |
90% (35,77) |
0,1586 |
37,77 |
|
100 |
2,000 |
2,096 |
72 |
2% (3,71) |
0% (0,00) |
0% (0,00) |
35% (14,49) |
63% (19,44) |
0,1057 |
24,70 |
|
|
5.000 |
3,699 |
3,699 |
72 |
4% (3,47) |
0% (0,00) |
0% (0,00) |
49% (11,99) |
47% (11,76) |
0,0742 |
17,22 |
|
|
50.000 |
4,699 |
4,745 |
72 |
0% (0,00) |
0% (0,00) |
0% (0,00) |
59% (17,39) |
41% (14,45) |
0,0975 |
22,75 |
|
|
500.000 |
5,699 |
5,824 |
72 |
19% (7,91) |
0% (0,00) |
0% (0,00) |
24% (8,99) |
57% (13,89) |
0,0794 |
18,43 |
|
|
5.000.000 |
6,699 |
6,177 |
72 |
51% (20,74) |
0% (1,59) |
0% (0,00) |
9% (8,47) |
40% (18,27) |
0,1246 |
29,30 |
|
|
50.000.000 |
7,699 |
7,069 |
72 |
17% (13,08) |
0% (0,00) |
0% (0,00) |
0% (0,00) |
83% (29,26) |
0,1367 |
32,28 |
|
|
3 |
30 |
1,477 |
1,457 |
72 |
0% (0,00) |
0% (0,00) |
0% (0,00) |
34% (24,33) |
66% (34,06) |
0,1776 |
42,67 |
|
100 |
2,000 |
1,911 |
72 |
16% (13,76) |
0% (0,00) |
0% (0,00) |
27% (18,01) |
58% (26,79) |
0,1504 |
35,70 |
|
|
5.000 |
3,699 |
3,628 |
72 |
10% (6,12) |
0% (0,00) |
0% (0,00) |
18% (8,09) |
71% (16,06) |
0,0821 |
19,07 |
|
|
50.000 |
4,699 |
4,587 |
72 |
2% (2,23) |
0% (0,00) |
0% (0,00) |
55% (13,21) |
44% (11,85) |
0,0774 |
17,96 |
|
|
500.000 |
5,699 |
5,524 |
72 |
0% (0,00) |
0% (0,00) |
0% (0,00) |
44% (12,53) |
56% (14,30) |
0,0822 |
19,10 |
|
|
5.000.000 |
6,699 |
6,442 |
71 |
22% (11,89) |
0% (0,00) |
0% (0,00) |
0% (0,00) |
78% (22,66) |
0,1100 |
25,73 |
|
|
50.000.000 |
7,699 |
7,109 |
71 |
10% (13,36) |
0% (0,00) |
21% (19,65) |
0% (0,00) |
69% (35,94) |
0,1827 |
44,01 |
|
Nota: La tabla únicamente incluye resultados con carga viral detectable.
a Calculadas mediante el procedimiento SAS MIXED.
b Número de pruebas válidas con carga viral detectable.
c Cálculo realizado utilizando la variabilidad total del procedimiento SAS MIXED.
d CV (%) log normal = sqrt(10^[SD^2 × ln(10)] – 1) × 100.
CV (%) = porcentaje del coeficiente de variación; HCV = virus de la hepatitis C; N.º = número; ARN = ácido ribonucleico; SD = desviación estándar; sqrt = raíz cuadrada.
El PCN fue del 100% con pruebas del miembro negativo del panel en el cobas® 6800 System tal como se presenta en la Tabla 30 a continuación.
Tabla 30. Porcentaje de concordancia de negativos con el miembro negativo del panel (reproducibilidad) en el cobas® 6800 System
|
Concentración de ARN del HCV esperada |
N.º de pruebas |
Resultados positivos |
Resultados negativos |
Porcentaje de concordancia de negativosa |
IC del 95%b |
|
Negativa |
108 |
0 |
108 |
100,00 |
(96,64, 100,00) |
a Porcentaje de concordancia de negativos = (número de resultados negativos ÷ número total de pruebas válidas en el miembro negativo del panel) × 100.
b Calculado mediante el método de intervalo de confianza binomial exacto de Clopper-Pearson.
IC = intervalo de confianza; HCV = virus de la hepatitis C; N.º = número; ARN = ácido ribonucleico.
Comparación entre el cobas® 6800 System y el cobas® 8800 System - Variabilidad entre lotes y reproducibilidad: Se analizó un conjunto de muestras idénticas para estudiar la variabilidad entre lotes y la reproducibilidad de la prueba COBAS® HCV en el cobas® 8800 System. El rendimiento de ambos sistemas es comparable. En la Tabla 31 se enumera el rendimiento de la precisión conseguida en la sección de reproducibilidad del estudio para el cobas® 6800 System y el cobas® 8800 System a lo largo del intervalo lineal de la prueba COBAS® HCV.
Tabla 31. Comparación de la desviación estándar de la precisión de la concentración de ARN de HCV (log10 UI/ml) para los genotipos 1-3 en el cobas® 6800 System y el cobas® 8800 System (reproducibilidad)
|
Nivel de concentración (UI/mL) |
Desviación estándar de la precisióna (n.º de pruebasb) |
|||||
|
cobas® 6800 System |
cobas® 8800 System |
|||||
|
Genotipo 1 |
Genotipo 2 |
Genotipo 3 |
Genotipo 1 |
Genotipo 2 |
Genotipo 3 |
|
|
1,0E+01 ≤ X < 1,0E+02 |
0,24 (68) 0,16 (72) |
0,16 (72) |
0,18 (72) 0,15 (72) |
0,23 (47) 0,15 (47) |
0,14 (48) |
0,17 (47) 0,17 (48) |
|
1,0E+02 ≤ X < 1,0E+03 |
- |
0,11 (72) |
- |
- |
0,12 (48) |
- |
|
1,0E+03 ≤ X < 1,0E+04 |
0,14 (72) |
0,07 (72) |
0,08 (72) |
0,13 (48) |
0,07 (48) |
0,08 (48) |
|
1,0E+04 ≤ X < 1,0E+05 |
0,17 (72) |
0,10 (72) |
0,08 (72) |
0,11 (48) |
0,06 (48) |
0,08 (48) |
|
1,0E+05 ≤ X < 1,0E+06 |
0,15 (72) |
0,08 (72) |
0,08 (72) |
0,11 (48) |
0,07 (47) |
0,10 (48) |
|
1,0E+06 ≤ X < 1,0E+07 |
0,10 (72) |
0,12 (72) |
0,11 (71) |
0,09 (48) |
0,13 (48) |
0,11 (48) |
|
1,0E+07 ≤ X < 1,0E+08 |
0,22 (72) |
0,14 (72) |
0,18 (71) |
0,16 (48) |
0,10 (48) |
0,19 (48) |
Nota: La agrupación de las precisiones observadas en los niveles de concentración se basa en la media de los resultados de la prueba en la escala sin transformar (UI/mL). La tabla únicamente incluye resultados con carga viral detectable; SD = desviación estándar.
“-” significa que no hay ningún resultado aplicable a este nivel.
a Desviación estándar de la precisión en unidades log10.
b Número de pruebas válidas con carga viral detectable.
Utilidad clínica: El estudio se diseñó para evaluar la capacidad del ensayo para predecir el resultado clínico.
El plan de tratamiento 1 incluía cuatro regímenes de tratamiento con una combinación de compuestos de AAD con o sin peg-IFN/RBV. Los participantes estaban infectados por el genotipo 1 del HCV y mostraron una respuesta parcial o nula durante un ciclo anterior del tratamiento combinado de peg-IFN/RBV.
El plan de tratamiento 2 incluía sujetos infectados por el genotipo 2 o 3 que no habían recibido tratamiento previo y que recibieron un ciclo de tratamiento combinado peg-IFN/RBV.
Los ensayos con la prueba COBAS® HCV se realizaron en cuatro centros. Tres centros estaban equipados con un cobas® 6800 System. Dos centros estaban equipados con un cobas® 8800 System. Un centro realizó las pruebas en ambos cobas® 6800 y 8800 Systems. Se utilizaron tres lotes del kit en el estudio y cada muestra se analizó con un lote del kit.
En la Tabla 32 a continuación se muestran las características demográficas e iniciales de los sujetos cuyas muestras se analizaron en el cobas® 6800 System y el cobas® 8800 System. La mayoría de los sujetos fueron hombres de más de 40 años e infectados con el genotipo 1 del HCV. En el estudio participaron sujetos infectados por los genotipos 1, 2 y 3 del HCV. La infección por HCV con los genotipos 4, 5 y 6 es infrecuente en los EE.UU.
Tabla 32. Características demográficas e iniciales de los sujetos para los cobas® 6800 y cobas® 8800 Systems
|
Características |
cobas® 6800 System |
cobas® 8800 System |
||
|---|---|---|---|---|
|
Estadística |
Sujetos |
Estadística |
Sujetos |
|
|
Total |
N |
401 |
N |
353 |
|
Plan de tratamiento |
||||
|
1 |
n (%) |
307 (76,6%) |
n (%) |
287 (81,3%) |
|
2 |
n (%) |
94 (23,4%) |
n (%) |
66 (18,7%) |
|
Categoría de edad (años) |
||||
|
< 40 |
n (%) |
90 (22,4%) |
n (%) |
81 (22,9%) |
|
≥ 40 |
n (%) |
311 (77,6%) |
n (%) |
272 (77,1%) |
|
Edad (años) |
||||
|
Media ± SD |
49 ± 11,1 |
Media ± SD |
49 ± 11,2 |
|
|
Mediana |
52 |
Mediana |
52 |
|
|
Rango |
20-76 |
Rango |
20-71 |
|
|
Sexo |
||||
|
Hombres |
n (%) |
276 (68,8%) |
n (%) |
245 (69,4%) |
|
Mujeres |
n (%) |
125 (31,2%) |
n (%) |
108 (30,6%) |
|
Raza/Etnia |
||||
|
Asiático |
n (%) |
3 (0,7%) |
n (%) |
2 (0,6%) |
|
Afroamericano |
n (%) |
13 (3,2%) |
n (%) |
12 (3,4%) |
|
Blanco/Caucásico |
n (%) |
357 (89,0%) |
n (%) |
318 (90,1%) |
|
Otros |
n (%) |
28 (7,0%) |
n (%) |
21 (5,9%) |
|
Genotipo |
||||
|
1A |
n (%) |
174 (43,4%) |
n (%) |
159 (45,0%) |
|
1B |
n (%) |
133 (33,2%) |
n (%) |
128 (36,3%) |
|
Global 1 |
n (%) |
307 (76,6%) |
n (%) |
287 (81,3%) |
|
2 |
n (%) |
31 (7,7%) |
n (%) |
22 (6,2%) |
|
3 |
n (%) |
63 (15,7%) |
n (%) |
44 (12,5%) |
|
Global distinto de 1 |
n (%) |
94 (23,4%) |
n (%) |
66 (18,7%) |
|
ARN inicial de HCV (log10 UI/mL) |
||||
|
Media ± SD |
6,32 ± 0,58 |
Media ± SD |
6,33 ± 0,56 |
|
|
Mediana |
6,41 |
Mediana |
6,41 |
|
|
Rango |
2,57-7,52 |
Rango |
2,77-7,52 |
|
|
Categoría de ARN inicial de HCV |
||||
|
< 400.000 UI/mL |
n (%) |
36 (9,0%) |
n (%) |
32 (9,1%) |
|
≥ 400.000 UI/mL |
n (%) |
363 (90,5%) |
n (%) |
304 (86,1%) |
|
Ausente |
n (%) |
2 (0,5%) |
n (%) |
17 (4,8%) |
HCV = virus de la hepatitis C; ARN = ácido ribonucleico; SD = desviación estándar.
Predicción sobre la respuesta al tratamiento con antivirales: Las características de rendimiento del ensayo se han establecido para individuos tratados con determinados regímenes de AAD. No hay información disponible sobre el valor predictivo del ensayo con la aplicación de otros tratamientos combinados de AAD.
Definiciones:
• Carga viral (CV) en la semana 2 = ARN de HCV < LLoQ = LoD = 15 UI/mL en la semana 2 del tratamiento con antivirales
• CV en la semana 2: ARN de HCV < LoD = LLoQ de 15 UI/mL
• CV en la semana 4: ARN de HCV < LLoQ en la semana 4 del tratamiento con antivirales
• CV en la semana 8: ARN de HCV < LLoQ en la semana 8 del tratamiento con antivirales
• CV en la semana 12: o bien una caída de como mínimo 2 log10 en el nivel de ARN de HCV en comparación con el valor inicial o bien ARN de HCV < LLoQ en la semana 12 del tratamiento con antivirales
• CV en la semana 24 (fin del tratamiento[FDT]): ARN de HCV < LLoQ en la semana 24 del tratamiento con antivirales
• Respuesta virológica sostenida en la semana 12 (RVS12): ARN de HCV < LLoQ en la semana 12 tras la finalización del tratamiento con antivirales cuantificado con una prueba de ARN de HCV independiente
Valor predictivo de la respuesta virológica para el éxito del tratamiento con antivirales: En este estudio, el valor predictivo positivo (VPP) para la CV en la semana 4 para predecir la RVS12 fue del 78,1% (IC del 95%: del 72,7 al 82,8%) en sujetos con el genotipo 1 y del 84,7% (IC del 95%: del 73,5 al 91,8%) en sujetos con genotipos distintos de 1 (Tabla 33). Por lo tanto, la CV en la semana 4 cuantificada por la prueba COBAS® HCV fue un indicador de predicción de la RVS12.
Para el plan de tratamiento 1, como un representante de un régimen con AAD, una CV en la semana 12 o en la semana 24 con la prueba COBAS® HCV predice la RVS12 en sujetos con el genotipo 1, con un VVP del 77,0% y un VPP del 78,6%, respectivamente. La ausencia de CV en la semana 12 o en la semana 24 predicen valores predictivos negativos (VPN) sin respuesta del 87,5% y del 100%, respectivamente (Tabla 33). Un análisis adicional de la CV en la semana 2 para predecir la RVS12 arroja un VPP del 79,4%, pero un VPN bajo del 29,9%.
En el plan de tratamiento 2, una CV en la semana 12 con la prueba COBAS® HCV en los genotipos 2 y 3 fue predictiva de la RVS12, con un VPP del 75,3%. A causa de la excepcionalidad de la falta de respuesta, la ausencia de una CV en la semana 12 no es una medida útil del resultado en esta población. El VPN fue del 50% y el número de sujetos sin respuesta fue reducido en este estudio (Tabla 33).
De forma global, este estudio demostró la utilidad de la prueba COBAS® HCV y el valor continuado de la valoración de las respuestas de ARN de HCV en la semana 4, en la semana 12 y en la semana 24 en pacientes sometidos a un tratamiento para infección crónica por HCV.
Tabla 33. Probabilidad de conseguir una respuesta virológica sostenida (RVS12) con una respuesta virológica (< 15 UI/mL) en una visita específica del tratamiento en los cobas® 6800 System
|
VPP (%) |
VPN (%) |
O |
||||||
|
Plan de tratamiento |
Genotipo |
Visita durante el tratamiento |
Sujetos elegibles |
Estimación |
n/N |
Estimación |
n/N |
Estimación |
|
1 |
1 |
Semana 2 |
290 |
79,4 (71,5, 85,5) |
100/126 |
29,9 (23,4, 37,3) |
49/164 |
1,64 (0,95, 2,83) |
|
Semana 4 |
290 |
78,1 (72,7, 82,8) |
200/256 |
50,0 (34,1, 65,9) |
17/34 |
3,57 (1,71, 7,45) |
||
|
Semana 8 |
285 |
76,8 (71,5, 81,4) |
212/276 |
66,7 (35,4, 87,9) |
6/9 |
6,63 (1,61, 27,24) |
||
|
Semana 12 |
286 |
77,0 (71,7, 81,5) |
214/278 |
87,5 (52,9, 97,8) |
7/8 |
23,41 (2,83, 193,80) |
||
|
Semana 24 |
282 |
78,6 (73,4, 83,0) |
217/276 |
100,0 (61,0, 100,0) |
6/6 |
47,52 (2,64, 855,66) |
||
|
2 |
Distinto de 1 |
Semana 4 |
82 |
84,7 (73,5, 91,8) |
50/59 |
47,8 (29,2, 67,0) |
11/23 |
5,09 (1,72, 15,04) |
|
Semana 12 |
83 |
75,3 (64,9, 83,4) |
61/81 |
50,0 (9,5, 90,5) |
1/2 |
3,05 (0,18, 51,04) |
||
Notas: Valor predictivo positivo (VPP) = PV ÷ (PV + PF) o la probabilidad de obtener una RVS12 si el sujeto ha presentado una respuesta virológica en una visita específica. La RVS12 se consigue si el sujeto presenta ARN de HCV < 15 UI/mL a las 12 semanas de la última dosis.
Valor predictivo negativo (VPN) = NV ÷ (NF + NV) o la probabilidad de no obtener una RVS12 si el sujeto no ha presentado una respuesta virológica en una visita específica.
Odds ratio (OR) = (PV × NV) ÷ (PF × NF).
IC = intervalo de confianza; NF = negativo falso; PF = positivo falso; HCV = virus de la hepatitis C; RVS12 = respuesta virológica sostenida 12 semanas después de la última dosis; NV = negativo verdadero; PV = positivo verdadero.
Comparación entre el cobas® 6800 System y el cobas® 8800 System - Utilidad clínica: Se analizó un conjunto de muestras idénticas para estudiar la utilidad clínica de la prueba COBAS® HCV en el cobas® 8800 System. Los sistemas demostraron un rendimiento altamente correlacionado sin diferencias significativas. En la Ilustración 10 siguiente se muestra un gráfico de regresión de Deming de las CV (log10 UI/mL) superiores a 15 UI/mL en todos los puntos de tiempo aplicables del tratamiento.
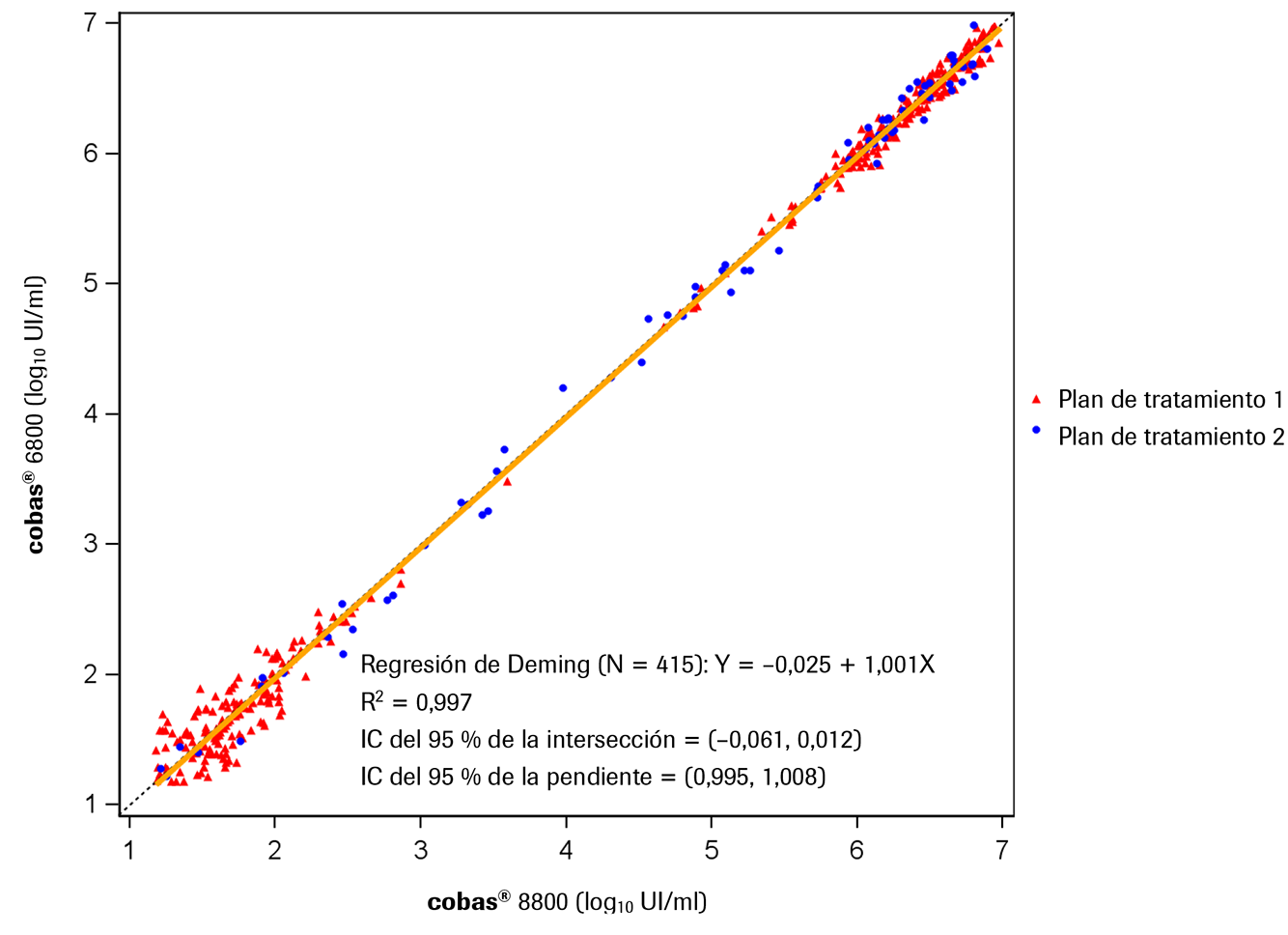
Ilustración 10. Gráfico de regresión lineal de Deming de las cargas virales (log10 UI/mL) al inicio, en la semana 2 y en la semana 4 (cobas® 6800 System frente a cobas® 8800 System)
Utilidad diagnóstica: El estudio se diseñó para evaluar la capacidad del ensayo para correlacionar correctamente el diagnóstico de sujetos positivos para anti-HCV con una infección activa por HCV.
En la Tabla 34 a continuación se muestran las características demográficas y clínicas de los sujetos cuyas muestras se analizaron en el cobas® 6800 System y el cobas® 8800 System.
Tabla 34. Características demográficas y clínicas por sistema (sujetos positivos para anticuerpos del HCV)
|
Características |
cobas® 6800 System |
cobas® 8800 System |
|---|---|---|
|
Total, n |
235 |
230 |
|
Condición clínica |
||
|
Positivo para anticuerpos del HCVa, n (%) |
||
|
Positivo para ARN del HCV |
154 (65,5%) |
150 (65,2%) |
|
Negativo para ARN del HCV |
81 (34,5%) |
80 (34,8%) |
|
Edad (años) |
||
|
Media ± SD |
48 ± 11,9 |
49 ± 11,9 |
|
Mediana |
50 |
50 |
|
Rango |
20-88 |
20-88 |
|
Sexo, n (%) |
||
|
Hombres |
132 (56,2%) |
127 (55,2%) |
|
Mujeres |
103 (43,8%) |
103 (44,8%) |
|
Raza, n (%) |
||
|
Negro/afroamericano |
49 (20,9%) |
48 (20,9%) |
|
Blanco/caucásico |
183 (77,9%) |
179 (77,8%) |
|
Otros |
3 (1,3%) |
3 (1,3%) |
|
Factor de riesgo, n (%) |
||
|
Solo generación baby boom (nacidos entre 1945-1965) |
114 (48,5%) |
112 (48,7%) |
|
Solo UDI |
22 (9,4%) |
22 (9,6%) |
|
Generación Baby boom y UDI |
23 (9,8%) |
22 (9,6%) |
|
No declarado, positivo para anticuerpos del HCV* |
76 (32,3%) |
74 (32,2%) |
a Para determinar el estado de ARN de HCV se utilizó el resultado de la prueba de HCV de VERSANT. En el caso de los sujetos cuyo resultado de la prueba de HCV de VERSANT no estaba disponible, se utilizó el resultado de la prueba de HCV de APTIMA. Cuando no se disponía de ninguno de los resultados de Versant y de Aptima, entonces se utilizó el resultado de la prueba COBAS® AMPLICOR® HCV, v2.0.
* La categoría “No declarado” incluye a los sujetos para los que faltan ambos factores de riesgo o son “No” o aquellos para los que falta un factor de riesgo y el otro presenta el valor “No”.
APTIMA = ensayo cualitativo de ARN de HCV de Aptima; HCV = virus de la hepatitis C; UDI = usuario de drogas inyectadas; SD = desviación estándar; VERSANT = ensayo cualitativo de ARN de HCV de VERSANT.
La sensibilidad de la prueba COBAS® HCV se evaluó en sujetos que habían estado expuestos previamente al HCV y habían obtenido un resultado positivo para anticuerpos del HCV en los cobas® 6800/8800 Systems (Tabla 35). La concordancia de la prueba COBAS® HCV con el estado de infección del paciente se determinó utilizando un valor de corte de < 25 UI/mL para definir la ausencia de una infección activa por HCV (Tabla 35).
Tabla 35. Concordancia de la prueba COBAS® HCV en el cobas® 6800 y el cobas® 8800 System con el estado de infección del paciente utilizando un valor de corte de 25 UI/mL
|
COBAS® HCV |
Estado de infección del paciente (PIS) |
|||||
|
cobas® 6800 System |
cobas® 8800 System |
|||||
|
Positivo para HCV |
Negativo para HCV |
Total |
Positivo para HCV |
Negativo para HCV |
Total |
|
|
ARN de HCV detectado superior a 25 UI/mL |
152 |
0 |
152 |
149 |
1 |
150 |
|
ARN de HCV no detectado o detectado inferior a 25 UI/mL |
0 |
81 |
81 |
0 |
79 |
79 |
|
Total |
152 |
81 |
233 |
149 |
80 |
229 |
|
Porcentaje de concordancia de positivos (IC con porcentaje del 95%) |
100,0% (97,5; 100,0) |
NA |
NA |
100,0% (97,5; 100,0) |
NA |
NA |
|
Porcentaje de concordancia de negativos (IC con porcentaje del 95%) |
NA |
100,0% (95,5; 100,0) |
NA |
NA |
98,8% (93,3; 99,8) |
NA |
Nota: En esta tabla solo se han incluido los resultados válidos de la prueba COBAS® HCV entre las muestras positivas para anticuerpos del HCV.
IC = intervalo de confianza; COBAS® HCV = COBAS® HCV para uso en los cobas® 6800/8800 Systems; HCV = virus de la hepatitis C; NA = no aplicable.
Este estudio demuestra la utilidad clínica de la prueba COBAS® HCV para diagnosticar correctamente sujetos con una infección activa en curso por ARN de HCV y para diferenciarlos de los sujetos con infecciones inactivas en una población con una exposición anterior al HCV (serología positiva a anticuerpos de HCV).
Reactividad cruzada en sujetos con enfermedad hepática no relacionada con el HCV: La reactividad cruzada de la prueba COBAS® HCV se evaluó con muestras que representaban una variedad de enfermedades hepáticas para las que una infección activa por HCV no era la causa subyacente. La prueba COBAS® HCV demostró la capacidad de determinar la ausencia de una infección activa por HCV en sujetos con una serie de enfermedades hepáticas por causas distintas al HCV (Tabla 36, Tabla 37, Tabla 38).
Tabla 36. Características demográficas y clínicas por sistema
|
Características |
cobas® 6800 System |
cobas® 8800 System |
|---|---|---|
|
Total, n |
247 |
181 |
|
Condición clínica |
||
|
Negativo para ARN del HCV, n (%) |
||
|
Enfermedad hepática alcohólica |
33 (13,4%) |
20 (11,0%) |
|
Hepatitis autoinmune |
37 (15,0%) |
32 (17,7%) |
|
HBV crónico |
30 (12,1%) |
30 (16,6%) |
|
Enfermedad de hígado graso |
66 (26,7%) |
38 (21,0%) |
|
Esteatohepatitis no alcohólica (EHNA) |
41 (16,6%) |
30 (16,6%) |
|
Cirrosis inespecífica |
6 (2,4%) |
3 (1,7%) |
|
Cirrosis biliar primaria |
33 (13,4%) |
28 (15,5%) |
|
Desconocidaa |
1 (0,4%) |
|
|
Edad (años) |
||
|
Media ± SD |
54 ± 13,1 |
54 ± 13,5 |
|
Mediana |
56 |
56 |
|
Rango |
20-81 |
20-81 |
|
Sexo, n (%) |
||
|
Hombres |
71 (28,7%) |
44 (24,3%) |
|
Mujeres |
104 (42,1%) |
74 (40,9%) |
|
Desconocido |
72 (29,1%) |
63 (34,8%) |
|
Raza, n (%) |
||
|
Asiático |
11 (4,5%) |
1 (0,6%) |
|
Negro/afroamericano |
13 (5,3%) |
11 (6,1%) |
|
Blanco/caucásico |
70 (28,3%) |
48 (26,5%) |
|
Otros |
7 (2,8%) |
1 (0,6%) |
|
Desconocido |
146 (59,1%) |
120 (66,3%) |
|
Generación Baby boom (nacidos entre 1945-1965), n (%) |
||
|
Sí |
80 (32,4%) |
63 (34,8%) |
|
No |
64 (25,9%) |
53 (29,3%) |
|
No declarado |
103 (41,7%) |
65 (35,9%) |
Tabla 37. Número de muestras negativas para ARN de HCV en el cobas® 6800 System con enfermedades hepáticas no relacionadas con el HCV dentro de las categorías de resultados de la prueba por condición clínica
|
Número de pruebas válidas |
|||||||
|
Condición clínica |
Diana no detectada |
< 1,50E+ |
1,50E+01 ≤ x < 2,50E+01 UI/mL |
2,50E+01 ≤ x ≤ 1,00E+08 UI/mL |
> 1,00E+08 UI/mL |
Total |
Especificidada % (IC del 95%)b |
|
Enfermedad hepática alcohólica |
33 |
0 |
0 |
0 |
0 |
33 |
100,0 (89,4, 100,0) |
|
Hepatitis autoinmune |
37 |
0 |
0 |
0 |
0 |
37 |
100,0 (90,5, 100,0) |
|
HBV crónico |
30 |
0 |
0 |
0 |
0 |
30 |
100,0 (88,4, 100,0) |
|
Enfermedad de hígado graso |
66 |
0 |
0 |
0 |
0 |
66 |
100,0 (94,6, 100,0) |
|
EHNA |
40 |
1* |
0 |
0 |
0 |
41 |
97,6 (87,1, 99,9) |
|
Cirrosis inespecífica |
6 |
0 |
0 |
0 |
0 |
6 |
100,0 (54,1, 100,0) |
|
Cirrosis biliar primaria |
33 |
0 |
0 |
0 |
0 |
33 |
100,0 (89,4, 100,0) |
|
Total |
245 |
1* |
0 |
0 |
0 |
246 |
99,6 (97,8, 100,0) |
Nota: En esta tabla solo se han incluido los resultados válidos de la prueba COBAS® HCV entre las muestras negativas para anticuerpos del HCV (enfermedad hepática no relacionada con el HCV). Se excluyó al único sujeto con esteatosis hepática.
a Especificidad clínica: porcentaje del número de resultados negativos de ARN con respecto al número total de muestras negativas para anticuerpos del HCV entre los resultados válidos de la prueba.
b IC del 95%: intervalo de confianza exacto del 95%.
* Muestra comunicada < LLOQ, ARN de HCV detectado a ~1,5 UI/mL.
IC = intervalo de confianza; HBV = virus de la hepatitis B; HCV = virus de la hepatitis C; EHNA = esteatohepatitis no alcohólica.
Tabla 38. Número de muestras negativas para ARN de HCV en el cobas® 8800 System con enfermedades hepáticas no relacionadas con el HCV dentro de las categorías de resultados de la prueba por condición clínica
|
Número de pruebas válidas |
|||||||
|
Condición clínica |
Diana no detectada |
< 1,50E+01 UI/mL |
1,50E+01 ≤ x < 2,50E+01 UI/mL |
2,50E+01 ≤ x |
> 1,00E+08 UI/mL |
Total |
Especificidada % (IC del 95%)b |
|
Enfermedad hepática alcohólica |
20 |
0 |
0 |
0 |
0 |
20 |
100,0 (83,2, 100,0) |
|
Hepatitis autoinmune |
32 |
0 |
0 |
0 |
0 |
32 |
100,0 (89,1, 100,0) |
|
HBV crónico |
30 |
0 |
0 |
0 |
0 |
30 |
100,0 (88,4, 100,0) |
|
Enfermedad de hígado graso |
38 |
0 |
0 |
0 |
0 |
38 |
100,0 (90,7, 100,0) |
|
EHNA |
30 |
0 |
0 |
0 |
0 |
30 |
100,0 (88,4, 100,0) |
|
Cirrosis inespecífica |
3 |
0 |
0 |
0 |
0 |
3 |
100,0 (29,2, 100,0) |
|
Cirrosis biliar primaria |
28 |
0 |
0 |
0 |
0 |
28 |
100,0 (87,7, 100,0) |
|
Total |
181 |
0 |
0 |
0 |
0 |
181 |
100,0 (98,0, 100,0) |
Nota: En esta tabla solo se han incluido los resultados válidos de la prueba COBAS® HCV entre las muestras negativas para anticuerpos del HCV (enfermedad hepática no relacionada con el HCV).
a Especificidad clínica: porcentaje del número de resultados negativos de ARN con respecto al número total de muestras negativas para anticuerpos del HCV entre los resultados válidos de la prueba.
b IC del 95%: intervalo de confianza exacto del 95%.
IC = intervalo de confianza; HBV = virus de la hepatitis B; HCV = virus de la hepatitis C; EHNA = esteatohepatitis no alcohólica.
Comparación entre el cobas® 6800 System y el cobas® 8800 System para el diagnóstico: Se analizó un subconjunto de muestras para confirmar la infección activa por HCV mediante la prueba COBAS® HCV en el cobas® 8800 System. La especificidad de la prueba COBAS® HCV en una variedad de enfermedades hepáticas en las que una infección activa por HCV no era la causa subyacente fue también del 100%. La concordancia de la prueba COBAS® HCV en el cobas® 8800 System con un estado de infección del paciente utilizando un valor de corte < 25 UI/mL para definir la ausencia de infección activa por HCV fue del 99,6%. Estos resultados indican que los cobas® 6800 y cobas® 8800 Systems son comparable en términos de diagnóstico del HCV activo mediante la prueba COBAS® HCV.
Conclusión: La prueba COBAS® HCV puede cuantificar el nivel de ARN de HCV para valorar el tratamiento y predecir la respuesta a un tratamiento con antivirales. Los resultados de este estudio demuestran la utilidad clínica de esta prueba para determinar la respuesta temprana al tratamiento para el control de los pacientes con infección crónica por HCV.
Asimismo, la prueba COBAS® HCV puede utilizarse como una ayuda en el diagnóstico de una infección activa por HCV en pacientes positivos para anticuerpos del HCV.
Equivalencia entre sistemas/comparación de sistemas: La equivalencia entre los cobas® 5800, cobas® 6800 y los cobas® 8800 Systems se demostró a partir de estudios de rendimiento. Los resultados incluidos en las Instrucciones de uso hacen patente la equivalencia de rendimiento entre todos los sistemas.
EVALUACIÓN NO CLÍNICA DEL RENDIMIENTO:
Características clave de rendimiento de los cobas® 6800/8800 Systems:
Límite de detección (LoD):
Estándar internacional de la OMS: El límite de detección de la prueba COBAS® HCV se determinó mediante el análisis de diluciones en serie del estándar internacional de la OMS para ARN del virus de la hepatitis C, en ensayos mediante tecnología de amplificación de ácidos nucleicos (cuarto estándar internacional de la OMS) para el genotipo 1a obtenido del National Institute for Biological Standards and Control (NIBSC), en suero y plasma conservado en EDTA humano negativo al HCV con volúmenes de procesamiento de muestras de 500 μL y 200 μL. El requisito de volumen de muestra mínimo era del 650 μL y de 350 μL respectivamente para el procesamiento en los cobas® 6800/8800 Systems. Se analizaron paneles con seis niveles de concentración, más uno negativo, para el volumen de procesamiento de muestras de 500 μL y con siete niveles de concentración para el volumen de procesamiento de muestras de 200 μL con tres lotes de reactivo de la prueba COBAS® HCV, con múltiples series analíticas, días, operadores e instrumentos.
Los resultados del suero y del plasma conservado en EDTA correspondientes a los dos volúmenes de procesamiento se detallan desde la Tabla 13 hasta la Tabla 16, respectivamente. El estudio demuestra que la prueba COBAS® HCV es capaz de detectar ARN del HCV en una concentración de 8,46 UI/mL con un intervalo de confianza del 95% de 7,50-9,79 UI/mL para el volumen de procesamiento de muestras de 500 μL en el caso de plasma conservado en EDTA, y en una concentración de 9,61 UI/mL con un intervalo de confianza del 95% de 8,70-10,95 UI/mL para el volumen de procesamiento de muestras de 500 μL en el caso de suero. Asimismo, este estudio también demuestra que la prueba COBAS® HCV es capaz de detectar ARN del HCV en una concentración de 24,93 UI/mL con un intervalo de confianza del 95% de 22,51-28,35 UI/mL para el volumen de procesamiento de muestras de 200 μL en el caso de plasma conservado en EDTA, y en una concentración de 33,25 UI/mL con un intervalo de confianza del 95% de 29,94-37,94 UI/mL para el volumen de procesamiento de muestras de 200 μL en el caso de suero. La diferencia entre plasma conservado en EDTA y suero con los volúmenes de procesamiento de muestras de 500 μL y 200 μL no fue estadísticamente significativa.
Tabla 13. Límite de detección en plasma conservado en EDTA (500 μL)
|
Concentración del título de entrada |
Número de réplicas válidas |
Número de positivos |
Tasa de posit. en% |
|
30 |
189 |
189 |
100,00 |
|
20 |
188 |
186 |
98,94 |
|
15 |
189 |
187 |
98,94 |
|
10 |
189 |
183 |
96,83 |
|
8 |
188 |
182 |
96,81 |
|
5 |
188 |
155 |
82,45 |
|
0 |
189 |
1* |
0,53 |
|
LoD según PROBIT con una tasa de positividad del 95% |
8,46 UI/mL Intervalo de confianza del 95%: 7,50-9,79 UI/mL |
||
* Muestras confirmadas como negativas por métodos analíticos alternativos.
Tabla 14. Límite de detección en suero (500 μL)
|
Concentración del título de entrada |
Número de réplicas válidas |
Número de positivos |
Tasa de posit. en% |
|
30 |
188 |
187 |
99,47 |
|
20 |
189 |
189 |
100,00 |
|
15 |
189 |
187 |
98,94 |
|
10 |
189 |
184 |
97,35 |
|
8 |
189 |
171 |
90,48 |
|
5 |
189 |
141 |
74,60 |
|
0 |
189 |
0 |
0,00 |
|
LoD según PROBIT con una tasa de positividad del 95% |
9,61 UI/mL Intervalo de confianza del 95%: 8,70-10,95 UI/mL |
||
Tabla 15. Límite de detección en plasma conservado en EDTA (200 μL)
|
Concentración del título de entrada |
Número de réplicas válidas |
Número de positivos |
Tasa de posit. en% |
|
80 |
189 |
189 |
100,00 |
|
60 |
189 |
189 |
100,00 |
|
50 |
188 |
187 |
99,47 |
|
40 |
189 |
185 |
97,88 |
|
25 |
189 |
179 |
94,71 |
|
20 |
189 |
177 |
93,65 |
|
12 |
188 |
136 |
72,34 |
|
0 |
189 |
1* |
0,53 |
|
LoD según PROBIT con una tasa de positividad del 95% |
24,93 UI/mL Intervalo de confianza del 95%: 22,51-28,35 UI/mL |
||
* Muestras confirmadas como negativas por métodos analíticos alternativos.
Tabla 16. Límite de detección en suero (200 μL)
|
Concentración del título de entrada |
Número de réplicas válidas |
Número de positivos |
Tasa de posit. en% |
|
80 |
189 |
189 |
100,00 |
|
60 |
189 |
188 |
99,47 |
|
50 |
189 |
186 |
98,41 |
|
40 |
189 |
184 |
97,35 |
|
25 |
189 |
167 |
88,36 |
|
20 |
189 |
156 |
82,54 |
|
12 |
189 |
125 |
66,14 |
|
0 |
189 |
0 |
0,00 |
|
LoD según PROBIT con una tasa de positividad del 95% |
33,25 UI/mL Intervalo de confianza del 95%: 29,94-37,94 UI/mL |
||
Intervalo lineal: El estudio de linealidad de la prueba COBAS® HCV se llevó a cabo con una serie de diluciones formadas por un panel de 16 muestras con el que se cubría el intervalo lineal esperado para el genotipo predominante (GT 1). Los miembros del panel con título alto se prepararon a partir de un stock de Armored RNA (arRNA) de título elevado mientras que los miembros del panel con título más bajo se prepararon a partir de una muestra clínica (MC). El panel de linealidad se diseñó para que existiera una superposición de títulos de aproximadamente 2 log10 entre los dos materiales de origen. El intervalo lineal esperado de la prueba COBAS® HCV se extiende desde el LLoQ (15 UI/mL para el volumen de procesamiento de 500 μL y 40 UI/mL para el volumen de procesamiento de 200 μL) hasta el ULoQ (1,00E+08 UI/mL en ambos volúmenes de procesamiento). El diseño del panel de linealidad permitía abarcar desde un concentración inferior al LLoQ (p. ej., 7,5 UI/mL) hasta una concentración superior al ULoQ (p. ej., 2,0E+08 UI/mL), así como incluir puntos de decisiones médicas. Asimismo, el panel de linealidad estaba diseñado para admitir parcialmente pasos de 1,0 log10 a lo largo del intervalo lineal. Para cada miembro del panel, se indicó la concentración nominal en UI/mL y el origen del ARN del HCV.
Para el volumen de procesamiento de 500 μL, la prueba COBAS® HCV es lineal para suero y plasma conservado en EDTA desde 15 UI/mL hasta 1,00E + 08 UI/mL, y presenta una desviación absoluta respecto al mejor ajuste de la regresión no lineal inferior a ± 0,24 log10. A lo largo del intervalo lineal, la exactitud de la prueba fue de ± 0,24 log10.
Para el volumen de procesamiento de 200 μL, la prueba COBAS® HCV es lineal para suero y plasma conservado en EDTA desde 40 UI/mL hasta 1,00E + 08 UI/mL, y presenta una desviación absoluta respecto al mejor ajuste de la regresión no lineal inferior a ± 0,24 log10. En el intervalo lineal, la exactitud de la prueba estuvo comprendida dentro de ± 0,24 log10 en plasma y ± 0,27 log10 en suero.
Consulte la Ilustración 4 a la Ilustración 7 para conocer los resultados representativos.
Ilustración 4. Linealidad en plasma conservado en EDTA (500 μL)
Ilustración 5. Linealidad en suero (500 μL)
Ilustración 6. Linealidad en plasma conservado en EDTA (200 μL).
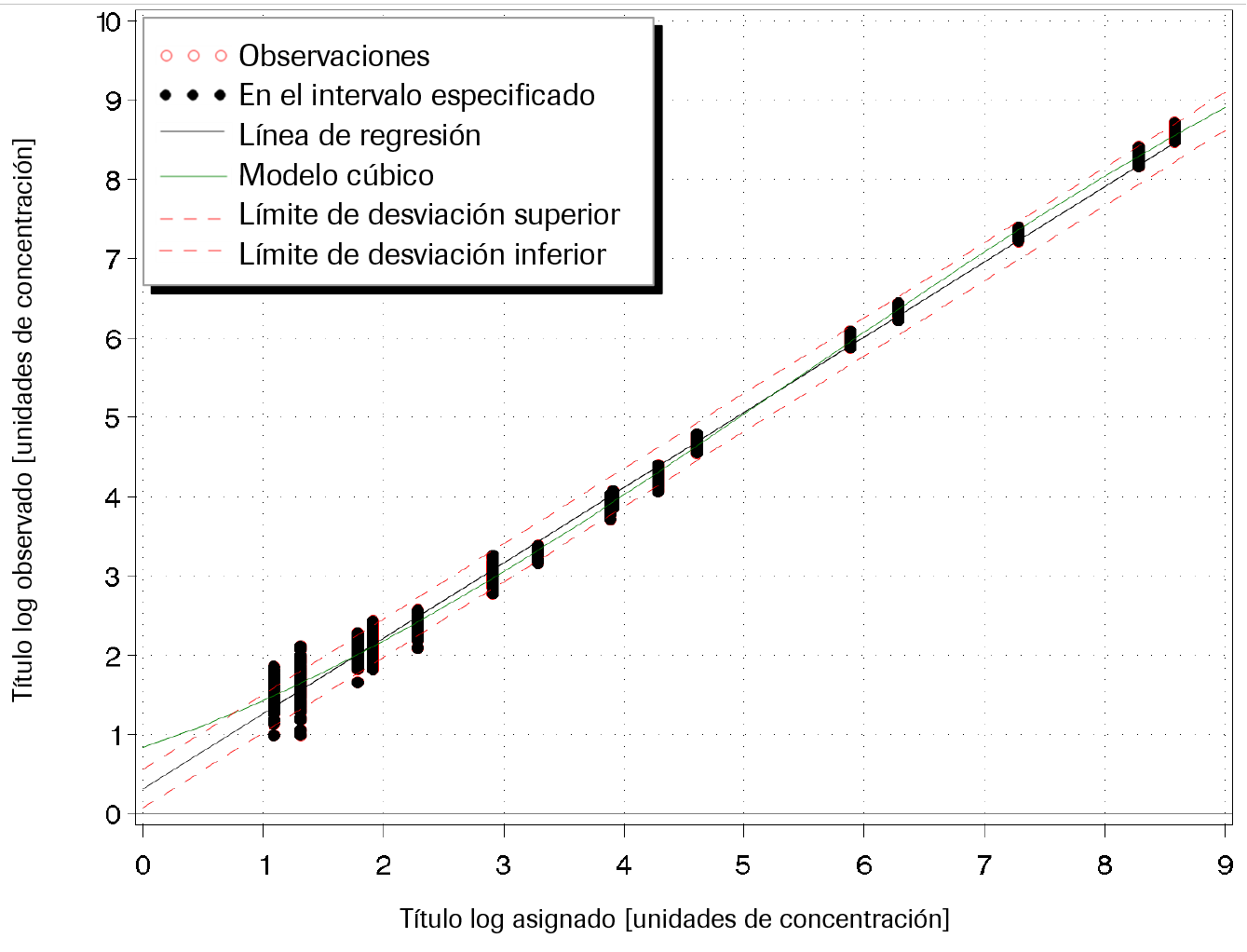
Ilustración 7. Linealidad en suero (200 μL).
Precisión (intralaboratorio): La precisión de la prueba COBAS® HCV se determinó mediante el análisis de diluciones en serie de muestras clínicas (MC) de HCV (genotipo 1) o de Armored RNA de HCV en suero o plasma conservado en EDTA negativos para el HCV. Se analizaron 13 niveles de dilución en plasma y 12 niveles en suero en dos réplicas para cada nivel en dos series analíticas durante 12 días, con un total de 48 réplicas por concentración. Cada muestra se sometió al procedimiento completo de la prueba COBAS® HCV en los cobas® 6800/8800 Systems totalmente automatizados. Por lo tanto, la precisión a la que se hace referencia en este documento engloba todas las fases del procedimiento de la prueba. El estudio se realizó con tres lotes de reactivos de la prueba COBAS® HCV. Los resultados se muestran de la Tabla 17 a la Tabla 20.
La prueba COBAS® HCV presentó una alta precisión para los tres lotes de reactivos analizados en un intervalo de concentración de 1,00E+01 UI/mL a 1,0E+07 UI/mL, con el volumen de procesamiento de muestras de 500 μL, y de 2,50E+01 UI/mL a 1,0E+07 UI/mL, con el volumen de procesamiento de muestras de 200 μL.
Tabla 17. Precisión intralaboratorio de la prueba COBAS® HCV (muestras de plasma conservado en EDTA – volumen de procesamiento de 500 μL)*
|
Concentración nominal (UI/mL) |
Concentración asignada (UI/mL) |
Material de origen |
Plasma conservado en EDTA |
|||
|
Lote 1 |
Lote 2 |
Lote 3 |
Todos los lotes |
|||
|
SD |
SD |
SD |
SD agrupada |
|||
|
1,00E+07 |
1,67E+07 |
arRNA |
0,04 |
0,05 |
0,03 |
0,04 |
|
1,00E+06 |
1,67E+06 |
arRNA |
0,05 |
0,05 |
0,06 |
0,05 |
|
4,00E+05 |
6,69E+05 |
arRNA |
0,03 |
0,04 |
0,05 |
0,04 |
|
5,00E+04 |
6,69E+04 |
MC |
0,08 |
0,06 |
0,06 |
0,06 |
|
1,00E+04 |
1,67E+04 |
arRNA |
0,05 |
0,05 |
0,04 |
0,05 |
|
1,00E+04 |
1,34E+04 |
MC |
0,03 |
0,06 |
0,05 |
0,05 |
|
4,00E+03 |
6,69E+03 |
arRNA |
0,05 |
0,06 |
0,06 |
0,06 |
|
1,00E+03 |
1,34E+03 |
MC |
0,05 |
0,06 |
0,05 |
0,05 |
|
1,00E+03 |
1,67E+03 |
arRNA |
0,05 |
0,07 |
0,05 |
0,06 |
|
1,00E+02 |
1,34E+02 |
MC |
0,06 |
0,09 |
0,05 |
0,07 |
|
1,00E+02 |
1,67E+02 |
arRNA |
0,10 |
0,06 |
0,06 |
0,08 |
|
5,00E+01 |
6,69E+01 |
MC |
0,09 |
0,17 |
0,10 |
0,13 |
|
1,00E+01 |
1,34E+01 |
MC |
0,26 |
0,21 |
0,13 |
0,21 |
* Se considera que los datos de título cuentan con una distribución log-normal y se analizan según la transformación de log10. Las columnas de desviaciones estándar (SD) presentan el total del título log-transformado para cada uno de los tres lotes de reactivo.
Tabla 18. Precisión intralaboratorio de la prueba COBAS® HCV (muestras de suero - volumen de procesamiento de 500 μL)*
|
Concentración nominal (UI/mL) |
Concentración asignada (UI/mL) |
Material de origen |
Suero |
|||
|
Lote 1 |
Lote 2 |
Lote 3 |
Todos los lotes |
|||
|
SD |
SD |
SD |
SD agrupada |
|||
|
1,00E+07 |
1,92E+07 |
arRNA |
0,03 |
0,07 |
0,04 |
0,05 |
|
1,00E+06 |
1,92E+06 |
arRNA |
0,05 |
0,06 |
0,04 |
0,05 |
|
4,00E+05 |
7,69E+05 |
arRNA |
0,03 |
0,07 |
0,03 |
0,05 |
|
5,00E+04 |
4,05E+04 |
MC |
0,07 |
0,06 |
0,04 |
0,06 |
|
1,00E+04 |
1,92E+04 |
arRNA |
0,06 |
0,06 |
0,04 |
0,05 |
|
1,00E+04 |
8,11E+03 |
MC |
0,05 |
0,06 |
0,04 |
0,05 |
|
4,00E+03 |
7,69E+03 |
arRNA |
0,04 |
0,08 |
0,04 |
0,06 |
|
1,00E+03 |
8,11E+02 |
MC |
0,05 |
0,06 |
0,06 |
0,05 |
|
1,00E+03 |
1,92E+03 |
arRNA |
0,06 |
0,05 |
0,05 |
0,05 |
|
1,00E+02 |
8,11E+01 |
MC |
0,10 |
0,18 |
0,10 |
0,13 |
|
1,00E+02 |
1,92E+02 |
arRNA |
0,07 |
0,08 |
0,09 |
0,08 |
|
5,00E+01 |
4,05E+01 |
MC |
0,09 |
0,14 |
0,18 |
0,14 |
* Se considera que los datos de título cuentan con una distribución log-normal y se analizan según la transformación de log10. Las columnas de desviaciones estándar (SD) presentan el total del título log-transformado para cada uno de los tres lotes de reactivo.
Tabla 19. Precisión intralaboratorio de la prueba COBAS® HCV (muestras de plasma conservado en EDTA - volumen de procesamiento de 200 μL)*
|
Concentración nominal (UI/mL) |
Concentración asignada (UI/mL) |
Material de origen |
Plasma conservado en EDTA |
|||
|
Lote 1 |
Lote 2 |
Lote 3 |
Todos los lotes |
|||
|
SD |
SD |
SD |
SD agrupada |
|||
|
1,00E+07 |
1,67E+07 |
arRNA |
0,04 |
0,06 |
0,05 |
0,05 |
|
1,00E+06 |
1,67E+06 |
arRNA |
0,04 |
0,03 |
0,05 |
0,04 |
|
4,00E+05 |
6,69E+05 |
arRNA |
0,04 |
0,06 |
0,03 |
0,04 |
|
5,00E+04 |
6,69E+04 |
MC |
0,05 |
0,06 |
0,05 |
0,06 |
|
1,00E+04 |
1,67E+04 |
arRNA |
0,05 |
0,05 |
0,05 |
0,05 |
|
1,00E+04 |
1,34E+04 |
MC |
0,07 |
0,06 |
0,05 |
0,06 |
|
4,00E+03 |
6,69E+03 |
arRNA |
0,05 |
0,06 |
0,05 |
0,05 |
|
1,00E+03 |
1,34E+03 |
MC |
0,08 |
0,08 |
0,06 |
0,07 |
|
1,00E+03 |
1,67E+03 |
arRNA |
0,04 |
0,07 |
0,05 |
0,05 |
|
1,00E+02 |
1,34E+02 |
MC |
0,11 |
0,15 |
0,13 |
0,13 |
|
1,00E+02 |
1,67E+02 |
arRNA |
0,10 |
0,10 |
0,13 |
0,11 |
|
7,50E+01 |
1,00E+02 |
MC |
0,15 |
0,12 |
0,11 |
0,13 |
|
2,50E+01 |
3,34E+01 |
MC |
0,19 |
0,20 |
0,22 |
0,21 |
* Se considera que los datos de título cuentan con una distribución log-normal y se analizan según la transformación de log10. Las columnas de desviaciones estándar (SD) presentan el total del título log-transformado para cada uno de los tres lotes de reactivo.
Tabla 20. Precisión intralaboratorio de la prueba COBAS® HCV (muestras de suero - volumen de procesamiento de 200 μL)*
|
Concentración nominal (UI/mL) |
Concentración asignada (UI/mL) |
Material de origen |
Suero |
|||
|
Lote 1 |
Lote 2 |
Lote 3 |
Todos los lotes |
|||
|
SD |
SD |
SD |
SD agrupada |
|||
|
1,00E+07 |
1,92E+07 |
arRNA |
0,02 |
0,06 |
0,03 |
0,04 |
|
1,00E+06 |
1,92E+06 |
arRNA |
0,03 |
0,06 |
0,04 |
0,04 |
|
4,00E+05 |
7,69E+05 |
arRNA |
0,04 |
0,09 |
0,04 |
0,06 |
|
5,00E+04 |
4,05E+04 |
MC |
0,05 |
0,06 |
0,06 |
0,06 |
|
1,00E+04 |
1,92E+04 |
arRNA |
0,05 |
0,07 |
0,04 |
0,06 |
|
1,00E+04 |
8,11E+03 |
MC |
0,04 |
0,05 |
0,05 |
0,05 |
|
4,00E+03 |
7,69E+03 |
arRNA |
0,04 |
0,07 |
0,04 |
0,05 |
|
1,00E+03 |
8,11E+02 |
MC |
0,10 |
0,09 |
0,08 |
0,09 |
|
1,00E+03 |
1,92E+03 |
arRNA |
0,05 |
0,07 |
0,04 |
0,05 |
|
1,00E+02 |
8,11E+01 |
MC |
0,17 |
0,30 |
0,17 |
0,22 |
|
1,00E+02 |
1,92E+02 |
arRNA |
0,13 |
0,13 |
0,09 |
0,12 |
|
7,50E+01 |
6,08E+01 |
MC |
0,11 |
0,16 |
0,12 |
0,13 |
* Se considera que los datos de título cuentan con una distribución log-normal y se analizan según la transformación de log10. Las columnas de desviaciones estándar (SD) presentan el total del título log-transformado para cada uno de los tres lotes de reactivo.
Verificación del genotipo: El rendimiento de la prueba COBAS® HCV para los genotipos del HCV se evaluó mediante:
• Determinación del límite de detección para los genotipos del 1b al 6 analizados con el volumen de procesamiento de muestras de 500 μL.
• Verificación del límite de detección para los genotipos del 1b al 6 analizados con el volumen de procesamiento de muestras de 200 μL.
• Verificación de la linealidad para los genotipos del 2 al 6.
Límite de detección para los genotipos del 1b al 6: El límite de detección de la prueba COBAS® HCV para los genotipos del 1b al 6 se determinó mediante el análisis de diluciones en serie de cada genotipo, en suero y plasma conservado en EDTA humanos y negativos para el HCV con el volumen de procesamiento de 500 μL. Se analizaron paneles con seis niveles de concentración más uno negativo con tres lotes de reactivos de la prueba COBAS® HCV, con múltiples series analíticas, días, operadores e instrumentos.
Los resultados para suero y plasma conservado en EDTA correspondientes al volumen de procesamiento de 500 μL se detallan en la Tabla 21 y la Tabla 22, respectivamente. El estudio demuestra que la prueba COBAS® HCV es capaz de detectar todos los genotipos del HCV analizados con un LoD similar al del genotipo 1a del HCV.
Tabla 21. Límite de detección para los genotipos de ARN del HCV en plasma conservado en EDTA (500 μL)
|
Genotipo |
LoD del 95% |
Intervalo de |
|
GT 1b |
11,32 UI/mL |
9,72-14,52 UI/mL |
|
GT 2 |
9,10 UI/mL |
7,83-11,80 UI/mL |
|
GT 3 |
8,68 UI/mL |
7,30-11,51 UI/mL |
|
GT 4 |
12,78 UI/mL |
10,69-17,20 UI/mL |
|
GT 5 |
11,63 UI/mL |
9,66-15,98 UI/mL |
|
GT 6 |
12,58 UI/mL |
9,78-20,10 UI/mL |
Tabla 22. Límite de detección para los genotipos de ARN del HCV en suero (500 μL)
|
Genotipo |
LoD del 95% |
Intervalo de |
|
GT 1b |
15,24 UI/mL |
12,40-21,58 UI/mL |
|
GT 2 |
12,51 UI/mL |
10,25-17,63 UI/mL |
|
GT 3 |
7,21 UI/mL |
6,10-9,50 UI/mL |
|
GT 4 |
11,62 UI/mL |
9,92-15,02 UI/mL |
|
GT 5 |
13,06 UI/mL |
10,64-18,68 UI/mL |
|
GT 6 |
11,15 UI/mL |
9,54-14,40 UI/mL |
Verificación del límite de detección para los genotipos del 1b al 6: Se diluyeron muestras clínicas de ARN del HCV para seis genotipos distintos (1b, 2, 3, 4, 5, 6) en tres niveles de concentración diferentes en suero y plasma conservado en EDTA. La determinación de la tasa de positividad se llevó a cabo con 63 réplicas para cada nivel. El análisis se realizó con tres lotes de reactivos de la prueba COBAS® HCV. En la Tabla 23 y en la Tabla 24 se muestran los resultados obtenidos con el suero y el plasma conservado en EDTA con un volumen de 200 μL. Estos resultados verifican que la prueba COBAS® HCV puede detectar ARN del HCV para los seis genotipos distintos en concentraciones de 33 UI/mL, con una tasa de positividad de ≥ 90,5% y un intervalo de confianza unilateral superior del 95% de ≥ 95,8%.
Tabla 23. Verificación del límite de detección de los genotipos de ARN del HCV en plasma conservado en EDTA (200 μL)
|
Genotipo |
17,5 UI/mL |
33 UI/mL |
50 UI/mL |
||||||
|
Número de réplicas válidas |
Número de positivos |
Tasa de posit. en% (IC del 95%*) |
Número de réplicas válidas |
Número de positivos |
Tasa de posit. en% (IC del 95%*) |
Número de réplicas válidas |
Número de positivos |
Tasa de posit. en% (IC del 95%*) |
|
|
1b |
63 |
50 |
79,4 |
63 |
61 |
96,8 |
63 |
63 |
100,0 |
|
2 |
63 |
51 |
81,0 |
63 |
62 |
98,4 |
63 |
62 |
98,4 |
|
3 |
63 |
56 |
89,0 |
63 |
58 |
92,1 |
63 |
63 |
100,0 |
|
4 |
63 |
54 |
85,7 |
63 |
57 |
90,5 |
63 |
63 |
100,0 |
|
5 |
63 |
57 |
90,5 |
63 |
61 |
96,8 |
63 |
63 |
100,0 |
|
6 |
63 |
47 |
74,6 |
63 |
57 |
90,5 |
63 |
62 |
98,4 |
* Intervalo de confianza unilateral superior del 95%
Tabla 24. Verificación del límite de detección para los genotipos de ARN del HCV en suero (200 μL)
|
Genotipo |
17,5 UI/mL |
33 UI/mL |
50 UI/mL |
||||||
|
Número de réplicas válidas |
Número de positivos |
Tasa de posit. en% (IC del 95%*) |
Número de réplicas válidas |
Número de positivos |
Tasa de posit. en% (IC del 95%*) |
Número de réplicas válidas |
Número de positivos |
Tasa de posit. en% (IC del 95%*) |
|
|
1b |
63 |
52 |
82,5 |
63 |
61 |
96,8 |
63 |
63 |
100,0 |
|
2 |
63 |
46 |
73,0 |
63 |
62 |
98,4 |
63 |
59 |
93,7 |
|
3 |
63 |
58 |
92,1 |
63 |
63 |
100,0 |
63 |
63 |
100,0 |
|
4 |
63 |
49 |
77,8 |
63 |
59 |
93,7 |
63 |
63 |
100,0 |
|
5 |
63 |
46 |
73,0 |
63 |
59 |
93,7 |
63 |
62 |
98,4 |
|
6 |
63 |
44 |
69,8 |
63 |
61 |
96,8 |
63 |
61 |
96,8 |
* Intervalo de confianza unilateral superior del 95%
Linealidad para los genotipos del 2 al 6: La serie de diluciones utilizada para la verificación del estudio de linealidad de los genotipos de la prueba COBAS® HCV consta de un panel de nueve miembros con el que se cubre el intervalo lineal esperado. Los miembros del panel con título alto se prepararon a partir de un stock de arRNA de título elevado mientras que los miembros del panel con título más bajo se prepararon a partir de una muestra clínica (MC) de título elevado. El panel de linealidad se diseñó para que existiera una superposición de títulos de aproximadamente 2 log10 entre los dos materiales de origen. El intervalo lineal de la prueba COBAS® HCV se extendía desde el LLoQ (15 UI/mL para un volumen de procesamiento de muestras de 500 μl, 40 UI/mL para un volumen de procesamiento de 200 μL) hasta el ULoQ (1,00E+08 UI/mL para ambos volúmenes de procesamiento) e incluía al menos un punto de decisión médica. El análisis se realizó con tres lotes de reactivos COBAS® HCV con los que se analizaron 15 réplicas por nivel en plasma conservado en EDTA.
La linealidad del intervalo lineal de la prueba COBAS® HCV se verificó para los cinco genotipos (2, 3, 4, 5 y 6). La desviación máxima entre la regresión lineal y el mejor ajuste de la regresión no lineal fue igual o menor que 0,24 log10.
Especificidad: La especificidad de la prueba COBAS® HCV se determinó mediante el análisis de muestras de suero y plasma conservado en EDTA negativas para el HCV obtenidas de donantes individuales. Se analizaron 300 muestras individuales de plasma conservado en EDTA y 300 muestras individuales de suero (600 resultados totales) con dos lotes de reactivos de la prueba COBAS® HCV. Todas las muestras dieron negativo para ARN del HCV. En el panel de la prueba, la especificidad de COBAS® HCV fue del 100% (límite de confianza del 95%: ≥ 99,5%).
Especificidad analítica: La especificidad analítica de la prueba COBAS® HCV se evaluó mediante la dilución de un panel de microorganismos en plasma conservado en EDTA positivo para ARN del HCV y negativo para ARN del HCV. Los microorganismos se añadieron al plasma humano conservado en EDTA normal negativo al virus y se analizaron con y sin ARN del HCV.
La prueba COBAS® HCV generó resultados negativos para todas las muestras de microorganismos sin diana del HCV y resultados positivos para todas las muestras de microorganismos con diana del HCV. Además, el título log10 medio de cada una de las muestras positivas para el HCV con organismos de posible reacción cruzada se situó en ± 0,3 log10 del título log10 medio del control positivo respectivo añadido.
Tabla 25. Microorganismos analizados para reactividad cruzada
|
Virus |
Bacterias |
Levadura |
|
|
Adenovirus tipo 5 |
Virus del Nilo Occidental |
Propionibacterium acnes |
Candida albicans |
|
Citomegalovirus |
Virus de la encefalitis de St. Louis |
Staphylococcus aureus |
|
|
Virus de Epstein-Barr |
Virus de la encefalitis del Valle Murray |
||
|
Virus de la hepatitis A |
Virus del dengue tipos 1, 2, 3 y 4 |
||
|
Virus de la hepatitis B |
Virus FSME (cepa HYPR) |
||
|
Virus de la hepatitis D |
Virus de la fiebre amarilla |
||
|
Virus de inmunodeficiencia humana 1 |
Virus del herpes humano tipo 6 |
||
|
Virus linfotrópico de células T humanas tipos 1 y 2 |
Virus del herpes simple tipos 1 y 2 |
||
|
Virus del papiloma humano |
Virus de la gripe A |
||
|
Virus de la varicela zóster |
Virus Zika |
||
Especificidad analítica: sustancias interferentes: Se analizaron niveles elevados de triglicéridos (34,5 g/L), bilirrubina conjugada (0,25 g/L), bilirrubina no conjugada (0,25 g/L), albúmina (58,7 g/L), hemoglobina (2,9 g/L) y ADN humano (2 mg/L) en muestras en presencia y ausencia de ARN del HCV. El análisis de las sustancias interferentes endógenas demostró que no interferían con el rendimiento de la prueba COBAS® HCV.
Asimismo, se analizó la presencia de enfermedades autoinmunes como lupus eritematoso sistémico (LES), factor reumatoide (FR) y anticuerpos antinucleares (ANA).
Con respecto a la sensibilidad, en el caso de dos donantes de LES, uno de FR y cuatro de ANA, las muestras individuales mostraron interferencias con la prueba COBAS® HCV. Una investigación de la causa raíz demostró que la prueba superaba la interferencia procedente de las muestras de donantes de LES y FR afectadas cuando se analizaba en presencia de 75 UI/mL de ARN del HCV.
Las cuatro muestras de donantes de ANA que presentaban interferencias con la prueba COBAS® HCV cuando se analizaron con 50 UI/mL de ARN del HCV seguían produciendo interferencias cuando se analizaron con 75 UI/mL de ARN del HCV. Para valorar si la interferencia observada era específica de los ANA, o específica del donante, se analizaron 15 donantes de ANA adicionales en presencia de 50 UI/mL y de 75 UI/mL de ARN del HCV. Ninguno de los donantes adicionales presentó interferencia alguna con la prueba COBAS® HCV, para ambas concentraciones analizadas, con respecto a la sensibilidad/cuantificación.
También se analizaron los compuestos farmacológicos de la Tabla 26 con una concentración tres veces superior a la Cmáx. Se demostró que ninguno de los compuestos farmacológicos analizados interferían con la especificidad y la cuantificación del ARN del HCV mediante la prueba COBAS® HCV.
Se ha demostrado que ninguna de las posibles sustancias interferentes afecta al rendimiento de la prueba. La prueba COBAS® HCV generó resultados negativos para todas las muestras sin diana del HCV, y resultados positivos para todas las muestras con diana del HCV. Además, el título log10 medio de cada una de las muestras positivas para el HCV con sustancias potencialmente interferentes se situó en ± 0,3 log10 del título log10 medio del control positivo respectivo añadido.
Tabla 26. Compuestos farmacológicos analizados para la interferencia con la cuantificación de ARN de HCV mediante la prueba COBAS® HCV
|
Clase de fármaco |
Nombre genérico del fármaco |
|
|
Moduladores del sistema inmunológico |
Peginterferón α-2a Peginterferón α-2b Ribavirina |
|
|
Inhibidor de la entrada del HIV |
Maraviroc |
|
|
Inhibidor de la integrasa del HIV |
Elvitegravir/cobicistat |
Raltegravir |
|
Inhibidor de la transcriptasa inversa no nucleósido del HIV |
Efavirenz |
Nevirapina |
|
Etravirina |
Rilpivirina |
|
|
Inhibidor de la proteasa del HIV |
Atazanavir |
Lopinavir |
|
Tipranavir |
Nelfinavir |
|
|
Darunavir |
Ritonavir |
|
|
Fosamprenavir |
Saquinavir |
|
|
Inhibidor de la proteasa del HCV |
Boceprevir |
Telaprevir |
|
Simeprevir |
||
|
Inhibidor de la transcriptasa inversa o de la polimerasa de ADN |
Abacavir |
Tenofovir |
|
Emtricitabina |
Adefovir dipivoxil |
|
|
Entecavir |
Zidovudina |
|
|
Foscarnet |
Aciclovir |
|
|
Cidofovir |
Valganciclovir |
|
|
Lamivudina |
Ganciclovir |
|
|
Telbivudina |
Sofosbuvir |
|
|
Compuestos para el tratamiento de infecciones oportunistas |
Azitromicina |
Pirazinamida |
|
Claritromicina |
Rifabutina |
|
|
Etambutol |
Rifampicina |
|
|
Fluconazol |
Sulfametoxazol |
|
|
Isoniazida |
Trimetoprima |
|
Correlación de métodos:
Evaluación del rendimiento de la prueba COBAS® HCV en comparación con la prueba COBAS® AmpliPrep/COBAS® TaqMan® HCV Quantitative, v2.0: Se comparó el rendimiento de la prueba COBAS® HCV y la prueba COBAS® AmpliPrep/COBAS® TaqMan® HCV Quantitative, v2.0 (TaqMan® HCV v2.0) mediante el análisis de muestras de suero y plasma conservado en EDTA procedentes de pacientes infectados con el HCV. Un total de 149 muestras de plasma conservado en EDTA y 122 muestras de suero de todos los genotipos del HCV, analizadas por duplicado, resultaron válidas y se encontraban dentro del intervalo de cuantificación de ambas pruebas. Se realizó el análisis de la regresión de Deming. La desviación del título medio de las muestras analizadas con las dos pruebas fue de 0,02 log10 (intervalo de confianza del 95%: 0,00; 0,04).
En la Ilustración 8 se muestran los resultados de la regresión de Deming. El símbolo * de la Ilustración 8 indica una única determinación.
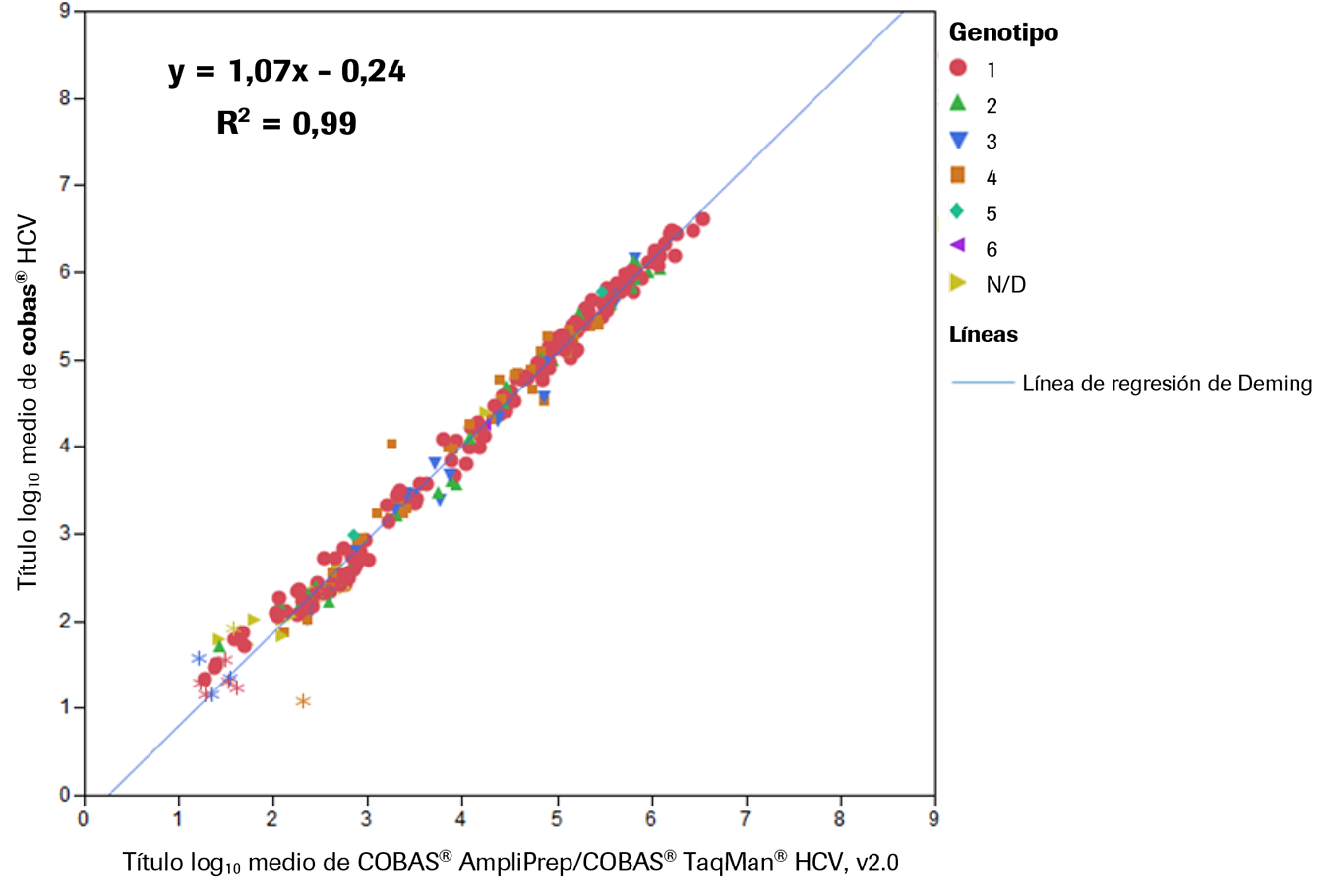
Ilustración 8. Comparación del análisis de regresión entre las pruebas COBAS® HCV y TaqMan® HCV, v2.0 en muestras de suero y plasma conservado en EDTA
Equivalencia de matrices: plasma conservado en EDTA frente a suero: Se analizaron 190 muestras de plasma conservado en EDTA y suero emparejadas para determinar la equivalencia de matrices. De ellas, 73 muestras emparejadas resultaron positivas para el HCV. Las muestras positivas para el HCV cubrían los genotipos del 1 al 4 en todo el intervalo lineal.
La desviación del título medio medida para las muestras de plasma conservado en EDTA y de suero emparejadas fue de -0,13 log10 (intervalo de confianza del 95%: -0,19; -0,07) (Ilustración 9).
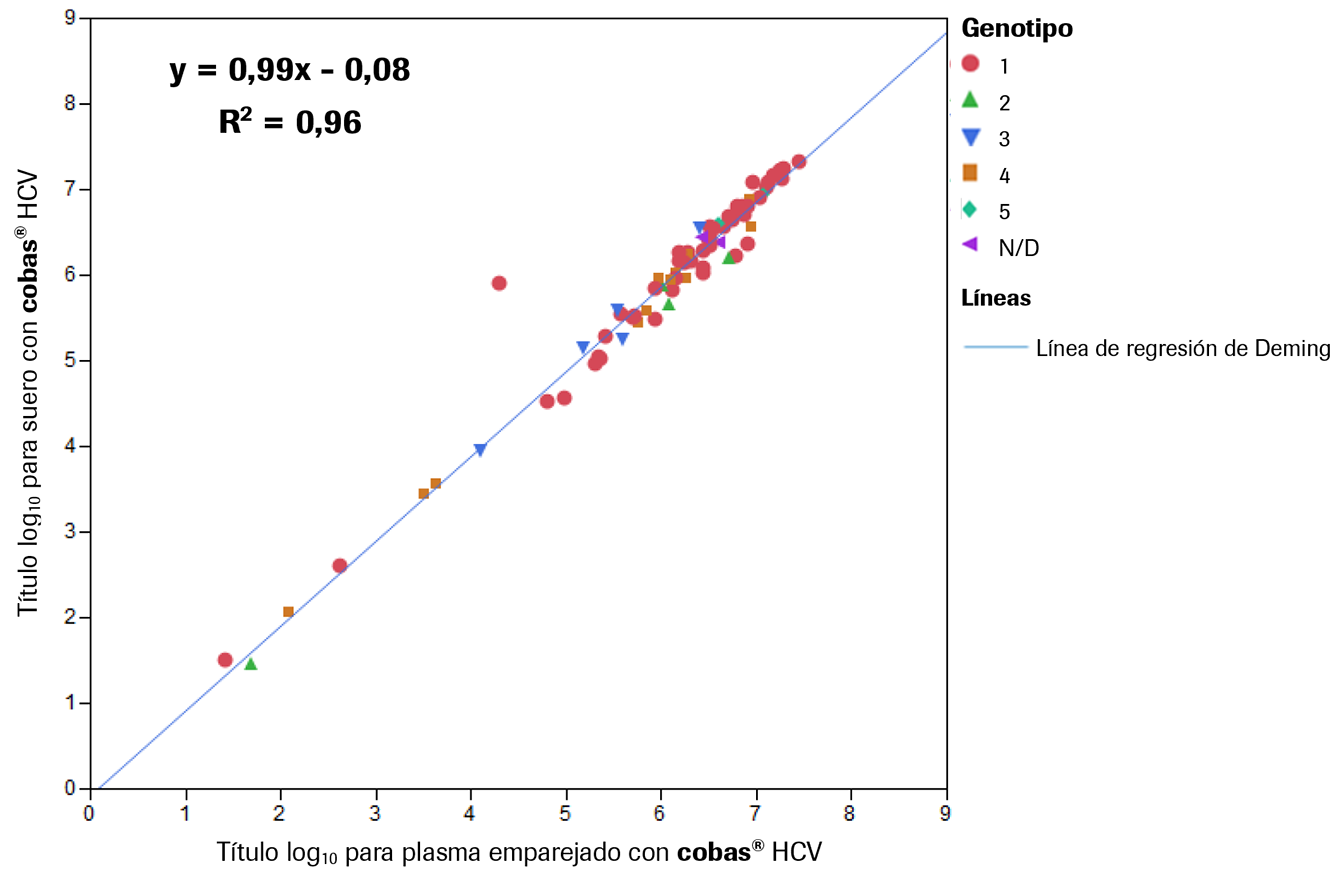
Ilustración 9. Rendimiento de la equivalencia de matrices entre muestras de plasma conservado en EDTA y suero
Fallo de todo el sistema:
La tasa de fallo de todo el sistema para la prueba COBAS® HCV se determinó mediante el análisis de 100 réplicas de plasma conservado en EDTA y 100 réplicas de suero a las que se añadió diana del HCV. Estas muestras se analizaron con una concentración de la diana de aproximadamente 3 × LoD.
Los resultados del estudio indican que todas las réplicas fueron válidas y positivas para el HCV, lo que representa una tasa de fallo de todo el sistema del 0%. El intervalo de confianza bilateral exacto del 95% fue del 0% para el límite inferior y del 3,62% para el límite superior para cada matriz [0%: 3,62%].
Contaminación por arrastre:
La tasa de contaminación por arrastre de la prueba COBAS® HCV se determinó mediante el análisis de 240 réplicas de una muestra de plasma humano normal conservado en EDTA negativa al virus (HIV, HCV y HBV) y de 225 réplicas de una muestra de HCV con un título alto de 4,0E+07 UI/ml. En total, se realizaron cinco series con muestras positivas y negativas utilizando un método de ensayo con configuración de tablero de ajedrez.
239 de las 240 réplicas de las muestras negativas resultaron válidas y se detectaron como negativas, por lo que la tasa de contaminación por arrastre fue del 0,42%. El intervalo de confianza bilateral exacto del 95% fue del 0,01% para el límite inferior, y del 2,3% para el límite superior (0%: 2,3%).
USO PREVISTO:
COBAS® HCV es una prueba de amplificación de ácidos nucleicos in vitro para la detección y la cuantificación del ARN de la hepatitis C (VHC), genotipos 1 a 6, en plasma conservado en EDTA o suero de pacientes infectados con el VHC.
COBAS® HCV se ha concebido como complemento en el diagnóstico de la infección del VHC en las poblaciones siguientes: pacientes que presentan evidencias de anticuerpos del VHC con evidencias de hepatopatía, pacientes de los que se sospecha que están infectados activamente con evidencias de anticuerpos del VHC y pacientes en riesgo de infección por VHC con anticuerpos del VHC. La detección de ARN del HCV indica que el virus se reproduce y, por lo tanto, constituye una evidencia de infección activa.
La prueba está diseñada para su uso en el tratamiento de pacientes con infección crónica por HCV en combinación con marcadores de infección clínicos y de laboratorio. La prueba puede utilizarse para predecir la probabilidad de una respuesta virológica sostenida (RVS) de forma temprana durante el curso de un tratamiento antiviral y para evaluar la respuesta viral a un tratamiento antiviral (terapia guiada para la respuesta) a través de la determinación de cambios en los niveles de ARN de VHC en suero o plasma conservado en EDTA. Los resultados deben interpretarse en el contexto de todos los resultados clínicos y de laboratorio relevantes.
INSTRUCCIONES DE USO:
Notas sobre el procedimiento:
• No utilice los reactivos de la prueba COBAS® HCV, el cobas® HBV/HCV/HIV-1 Control Kit, el cobas® NHP Negative Control Kit ni los reactivos cobas omni después de la fecha de caducidad.
• No reutilice el material fungible. Son de un solo uso.
• Consulte la Asistencia al usuario o la Guía del usuario del cobas® 5800 System o los cobas® 6800/8800 Systems para obtener información sobre el correcto mantenimiento de los instrumentos.
Ejecución de la prueba COBAS® HCV en el cobas® 5800 System: La prueba COBAS® HCV puede ejecutarse con dos volúmenes de muestra obligatorios: 350 μL (para el flujo de trabajo de muestras de 200 μL) y 650 μL (para el flujo de trabajo de muestras de 500 μL). El procedimiento de la prueba se describe con detalle en la Asistencia al usuario o la Guía del usuario del cobas® 5800 System. La Ilustración 2 siguiente resume el procedimiento.
• Nota: No utilice el flujo de trabajo de muestras de 200 μl si se espera que la carga viral sea ≤ 100 UI/mL. Debe procurarse un volumen de sangre suficiente para permitir el uso del volumen de procesamiento preferido para suero o plasma conservado en EDTA de 500 μL (para un requisito de volumen de muestra mínimo total de 650 μL).
Ilustración 2. Procedimiento de la prueba COBAS® HCV en el cobas® 5800 System
|
1 |
Inicie una sesión en el sistema |
|
2 |
Cargue las muestras en el sistema: • Cargue los racks de muestras en el sistema • El sistema se prepara automáticamente • Solicite las pruebas |
|
3 |
Cargue los reactivos y el material fungible según las indicaciones del sistema: • Cargue el casete de reactivo específico de la prueba • Cargue los mini racks de control • Cargue las puntas de procesamiento • Cargue las puntas de elución • Cargue las placas de procesamiento • Cargue las placas de residuos líquidos • Cargue las placas de amplificación • Cargue el casete de MGP • Cargue el diluyente de muestras • Cargue el reactivo de lisis • Cargue el reactivo de lavado |
|
4 |
Seleccione el botón de inicio de procesamiento en la interfaz de usuario para iniciar la serie analítica. Las series siguientes se iniciarán de forma automática si no se posponen manualmente |
|
5 |
Revise y exporte los resultados |
|
6 |
Retire y tape todos los tubos de muestra que cumplan los requisitos de volumen mínimo para utilizarlos en el futuro, en caso necesario. Limpieza del instrumento: • Descargue los mini racks de control vacíos • Descargue el casete de reactivo específico de la prueba vacío • Vacíe el cajón de placas de amplificación • Vacíe los residuos líquidos • Vacíe los residuos sólidos |
Ejecución de la prueba COBAS® HCV en los cobas® 6800/8800 Systems: La prueba COBAS® HCV puede ejecutarse con dos volúmenes de muestra obligatorios: 350 μL (para el flujo de trabajo de muestras de 200 μL) y 650 μL (para el flujo de trabajo de muestras de 500 μL). El procedimiento de la prueba se describe con detalle en la Asistencia al usuario o la Guía del usuario de los cobas® 6800/8800 Systems. La Ilustración 3 siguiente resume el procedimiento.
• Nota: No utilice el flujo de trabajo de muestras de 200 μL si se espera que la carga viral sea ≤ 100 UI/mL. Debe procurarse un volumen de sangre suficiente para permitir el uso del volumen de procesamiento preferido para suero o plasma conservado en EDTA de 500 μL (para un requisito de volumen de muestra mínimo total de 650 μL).
Ilustración 3. Procedimiento de la prueba COBAS® HCV en los cobas® 6800/8800 Systems
|
1 |
Inicie una sesión en el sistema Pulse el botón “Iniciar” para preparar el sistema Solicite las pruebas |
|
2 |
Cargue los reactivos y el material fungible según las indicaciones del sistema • Cargue el casete de reactivo específico de la prueba • Cargue los casetes de control • Cargue las puntas de pipeta • Cargue las placas de procesamiento • Cargue el reactivo MGP • Cargue las placas de amplificación • Cargue el diluyente de muestras • Cargue el reactivo de lisis • Cargue el reactivo de lavado |
|
3 |
Cargue las muestras en el sistema • Cargue los racks de muestras y los racks para puntas obstruidas en el módulo de suministro de muestras • Confirme que las muestras han sido aceptadas en el módulo de transferencia |
|
4 |
Inicie la serie analítica mediante el botón “Iniciar manualmente” de la interfaz de usuario o ejecute un inicio automático después de 120 minutos, o cuando la serie esté llena |
|
5 |
Revise y exporte los resultados |
|
6 |
Retire y tape todos los tubos de muestra que cumplan los requisitos de volumen mínimo para utilizarlos en el futuro, en caso necesario Limpie el instrumento • Descargue los casetes de control vacíos • Vacíe el cajón de placas de amplificación • Vacíe los residuos líquidos • Vacíe los residuos sólidos |
INFORMACIÓN ADICIONAL:
Características principales de la prueba:
|
Tipo de muestra |
Plasma conservado en EDTA, suero |
|
Cantidad mínima de muestra necesaria |
650 μL o 350 μL |
|
Volumen de procesamiento de muestras |
500 μL o 200 μL |
|
Sensibilidad analítica |
15 UI/mL (500 μL) 40 UI/mL (200 μL) |
|
Intervalo lineal |
500 μL: 15 UI/mL-1,0E+08 UI/mL |
|
200 μL: 40 UI/mL-1,0E+08 UI/mL |
|
|
Especificidad |
100% (intervalo de confianza unilateral del 95%: 99,5%) |
|
Genotipos detectados |
Genotipos 1-6 del HCV |
Símbolos:
Los símbolos siguientes se emplean en el rotulado de todos los productos de diagnóstico por PCR de Roche.
Tabla 39. Símbolos utilizados en las etiquetas de los productos de PCR para diagnóstico de Roche

Bibliografía:
1. Farci P. Choo QL, Kuo G, Weiner AJ, Overby LR, Bradley DW, Houghton M. Isolation of a cDNA clone derived from a blood-borne non-A, non-B viral hepatitis genome [Science 1989;244:359-362]. J Hepatol. 2002;36:582-5. PMID: 11983439.
2. Armstrong GL, Wasley A, Simard EP, et al. The prevalence of hepatitis C virus infection in the United States, 1999 through 2002. Ann Intern Med. 2006;144:705-14. PMID: 16702586.
3. Rustgi VK. The epidemiology of hepatitis C infection in the United States. J Gastroenterol. 2007;42:513-21. PMID: 17653645.
4. Lauer GM, Walker BD. Hepatitis C virus infection. N Engl J Med. 2001;345:41-52. PMID: 11439948.
5. McHutchison JG, Gordon SC, Schiff ER, et al. Interferon alfa-2b alone or in combination with ribavirin as initial treatment for chronic hepatitis C. Hepatitis Interventional Therapy Group. N Engl J Med. 1998;339:1485-92. PMID: 9819446.
6. Davis GL, Esteban-Mur R, Rustgi V, et al. Interferon alfa-2b alone or in combination with ribavirin for the treatment of relapse of chronic hepatitis C. International Hepatitis Interventional Therapy Group. N Engl J Med. 1998;339:1493-9. PMID: 9819447.
7. Manns MP, McHutchison JG, Gordon SC, et al. Peginterferon alfa-2b plus ribavirin compared with interferon alfa-2b plus ribavirin for initial treatment of chronic hepatitis C: a randomised trial. Lancet. 2001;358:958-65. PMID: 11583749.
8. Fried MW, Shiffman ML, Reddy KR, et al. Peginterferon alfa-2a plus ribavirin for chronic hepatitis C virus infection. N Engl J Med. 2002;347:975-82. PMID: 12324553.
9. Hadziyannis SJ, Sette H, Jr., Morgan TR, et al. Peginterferon-alpha2a and ribavirin combination therapy in chronic hepatitis C: a randomized study of treatment duration and ribavirin dose. Ann Intern Med. 2004;140:346-55. PMID: 14996676.
10. Ghany MG, Strader DB, Thomas DL, Seeff LB. Diagnosis, management, and treatment of hepatitis C: an update. Hepatology. 2009;49:1335-74. PMID: 19330875.
11. Ghany MG, Nelson DR, Strader DB, Thomas DL, Seeff LB. An update on treatment of genotype 1 chronic hepatitis C virus infection: 2011 practice guideline by the American Association for the Study of Liver Diseases. Hepatology. 2011;54:1433-44. PMID: 21898493.
12. Poordad F, McCone J, Jr., Bacon BR, et al. Boceprevir for untreated chronic HCV genotype 1 infection. N Engl J Med. 2011;364:1195-206. PMID: 21449783.
13. Jacobson IM, McHutchison JG, Dusheiko G, et al. Telaprevir for previously untreated chronic hepatitis C virus infection. N Engl J Med. 2011;364:2405-16. PMID: 21696307.
14. Bacon BR, Gordon SC, Lawitz E, et al. Boceprevir for previously treated chronic HCV genotype 1 infection. N Engl J Med. 2011;364:1207-17. PMID: 21449784.
15. Zeuzem S, Andreone P, Pol S, et al. Telaprevir for retreatment of HCV infection. N Engl J Med. 2011;364:2417-28. PMID: 21696308.
16. Lawitz E, Mangia A, Wyles D, et al. Sofosbuvir for previously untreated chronic hepatitis C infection. N Engl J Med. 2013;368:1878-87. PMID: 23607594.
17. Jacobson IM, Gordon SC, Kowdley KV, et al. Sofosbuvir for hepatitis C genotype 2 or 3 in patients without treatment options. N Engl J Med. 2013;368:1867-77. PMID: 23607593.
18. Liang TJ, Ghany MG. Current and future therapies for hepatitis C virus infection. N Engl J Med. 2013;368:1907-17. PMID: 23675659.
19. Rutter K, Hofer H, Beinhardt S, et al. Durability of SVR in chronic hepatitis C patients treated with peginterferon- α2a/ribavirin in combination with a direct-acting anti-viral. Aliment Pharmacol Ther. 2013;38:118-23. PMID: 23710895.
20. Longo MC, Berninger MS, Hartley JL. Use of uracil DNA glycosylase to control carry-over contamination in polymerase chain reactions. Gene. 1990;93:125-8. PMID: 2227421.
21. Savva R, McAuley-Hecht K, Brown T, Pearl L. The structural basis of specific base-excision repair by uracil-DNA glycosylase. Nature. 1995;373:487-93. PMID: 7845459.
22. Mol CD, Arvai AS, Slupphaug G, et al. Crystal structure and mutational analysis of human uracil-DNA glycosylase: structural basis for specificity and catalysis. Cell. 1995;80:869-78. PMID: 7697717.
23. Higuchi R, Dollinger G, Walsh PS, Griffith R. Simultaneous amplification and detection of specific DNA sequences. Biotechnology (N Y). 1992;10:413-7. PMID: 1368485.
24. Heid CA, Stevens J, Livak KJ, Williams PM. Real time quantitative PCR. Genome Res. 1996;6:986-94. PMID: 8908518.
25. Centers for Disease Control and Prevention. Biosafety in Microbiological and Biomedical Laboratories, 5th ed. https:// www.cdc.gov/labs/pdf/CDC-BiosafetyMicrobiologicalBiomedicalLaboratories-2009-P.PDF. Accessed December 2, 2020.
26. Clinical and Laboratory Standards Institute. Protection of laboratory workers from occupationally acquired infections. Approved Guideline-Fourth Edition. https://clsi.org/media/1459/m29a4_sample.pdf. Accessed December 2, 2020.
Asistencia técnica:
Para obtener asistencia técnica, póngase en contacto con su filial local: https://www.roche.com/about/business/roche_worldwide.htm
Fabricante e importador:
Tabla 40. Fabricante e importador
ROCHE MOLECULAR SYSTEMS, INC.
1080 US Highway 202 South
Branchburg, NJ 08876 USA
Fabricado en los EE. UU.
Roche Diagnostics GmbH
Sandhofer Strasse 116
68305 Mannheim, Germany
Marcas registradas y patentes
Consulte: https://diagnostics.roche.com/us/en/about-us/patents
Copyright
©2022 Roche Molecular Systems, Inc.
COBAS® HCV: P/N: 09040765190
Para uso en el sistema cobas® 5800
cobas® HBV/HCV/HIV-1 Control Kit: P/N: 09040773190
cobas® NHP Negative Control Kit: P/N: 09051554190
Para uso en sistemas cobas® 6800/8800
cobas® HBV/HCV/HIV-1 Control Kit:
P/N: 06997767190 o P/N: 09040773190
cobas® NHP Negative Control Kit
P/N: 07002220190 o P/N: 09051554190
RESUMEN Y EXPLICACIÓN DE LA PRUEBA:
Información de referencia: Se considera que el virus de la hepatitis C (HCV) es el agente etiológico principal responsable del 90 al 95% de los casos de hepatitis postransfusional.1-4 El HCV es un virus ARN monocatenario de sentido positivo con un genoma de aproximadamente 9.500 nucleótidos que codifican 3.000 aminoácidos. Como virus transportado en sangre, el HCV es transmisible a través de la sangre y los productos hemoderivados. La adopción más o menos generalizada de medidas de detección sistemática del HCV en sangre ha reducido de forma considerable el riesgo de hepatitis asociado a las transfusiones. La incidencia más alta de infección por HCV se asocia al abuso de drogas por vía intravenosa y, en menor medida, a otras exposiciones percutáneas.4
La determinación cuantitativa de ARN del HCV para la medición de cargas víricas de la línea de base y la monitorización del tratamiento está bien establecida para demostrar la eficacia de la respuesta antiviral al tratamiento combinado de interferón pegilado y ribavirina (pegIFN/RBV).5-9 Las directrices para la gestión y el tratamiento del HCV10,11 recomiendan realizar pruebas de determinación cuantitativa del ARN del HCV antes del inicio del tratamiento con antivirales, a intervalos de tiempo específicos durante el tratamiento (terapia guiada por la respuesta [TGR]) y a las 12 semanas o más tras la finalización del tratamiento.
El objetivo del tratamiento es la ausencia de ARN del HCV detectable mediante una prueba sensible realizada 12 semanas después de finalizar el tratamiento, lo que indicaría que se ha logrado una respuesta virológica sostenida (RVS).10
La determinación de la cinética viral durante el tratamiento se ha utilizado para personalizar aún más la duración del mismo gracias a los antivirales de acción directa (AAD) aprobados más recientemente, los inhibidores de la proteasa telaprevir y boceprevir.12-15
Motivos para el uso de la prueba de HCV: Ante la dinámica y extensa senda de fármacos descubiertos para futuros tratamientos del HCV, la monitorización de la carga viral continua siendo la principal prueba de laboratorio para confirmar que se ha conseguido la RVS con AAD, tales como los inhibidores de la proteasa de segunda generación, los inhibidores nucleósidos de la polimerasa del HCV y otros mecanismos de acción antiviral.16-19
En resumen, COBAS® HCV para uso con cobas® 5800/6800/8800 Systems es una prueba cuantitativa para la determinación de ARN del HCV y cinética viral para uso en laboratorios que realizan ensayos clínicos, y para el tratamiento de pacientes con HCV en la práctica clínica habitual.
Explicación de la prueba: COBAS® HCV es una prueba cuantitativa que se realiza en el cobas® 5800 System, el cobas® 6800 System o el cobas® 8800 System. La prueba COBAS® HCV permite la detección y cuantificación de ARN de HCV en plasma conservado en EDTA o suero de pacientes infectados. Se emplean sondas dobles que detectan y cuantifican, pero no discriminan, los genotipos 1-6. La cuantificación de la carga viral se realiza mediante un estándar de cuantificación de Armored RNA diferente del HCV (RNA-QS) que se añade a cada una de las muestras durante la preparación. El RNA-QS también actúa como control interno para monitorizar todo el proceso de preparación de las muestras y amplificación mediante PCR. Además, la prueba utiliza tres controles externos: uno positivo de título alto, uno positivo de título bajo y uno negativo. Los controles externos positivo alto y positivo bajo se fabrican mediante dilución a partir de material en stock con un título rastreable al estándar internacional de la ONU para el HCV. Cada lote de kits de amplificación/detección presenta una calibración rastreable al estándar internacional de la ONU para el HCV.
Principios del procedimiento: La prueba COBAS® HCV se basa en la preparación de muestras totalmente automática (extracción y purificación de ácidos nucleicos), seguida de un proceso de amplificación y detección mediante PCR. El cobas® 5800 System se ha diseñado como un único instrumento integrado. Los cobas® 6800/8800 Systems constan del módulo de suministro de muestras, el módulo de transferencia, el módulo de procesamiento y el módulo analítico. La gestión de datos automática se realiza mediante el software cobas® 5800 o cobas® 6800/8800 Systems, que asigna los resultados del análisis a todas las pruebas como diana no detectada, < LLoQ (límite de cuantificación inferior), > ULoQ (límite de cuantificación superior) o ARN de HCV detectado, un valor del intervalo lineal LLoQ < x < ULoQ. Los resultados pueden revisarse directamente en la pantalla del sistema, exportarse o imprimirse como informe PDF.
La extracción de ácidos nucleicos de las muestras de paciente, controles externos y moléculas de Armored RNA-QS añadido se realiza simultáneamente. En resumen, los ácidos nucleicos víricos se liberan al añadir proteinasa y reactivo de lisis a la muestra. Los ácidos nucleicos liberados se unen a la superficie de sílice de las partículas de vidrio magnéticas añadidas. Las sustancias sin unir y las impurezas, como las proteínas desnaturalizadas, los restos celulares y potenciales inhibidores de la PCR se eliminan en los siguientes pasos del buffer de lavado lavado, y los ácidos nucleicos purificados se eluyen de las partículas de vidrio magnéticas mediante el buffer de elución a temperatura elevada.
La amplificación selectiva de los ácidos nucleicos de la diana de la muestra de paciente se lleva a cabo mediante cebadores que van en un sentido y en sentido contrario específicos del virus diana, que se seleccionan de regiones altamente conservadas del HCV. La amplificación selectiva de RNA-QS se realiza mediante cebadores que van en un sentido y en sentido contrario específicos de la secuencia que se seleccionan de modo que no presenten homología alguna con el genoma del HCV. Para los procesos de transcripción inversa y amplificación mediante PCR se utiliza una enzima ADN polimerasa termoestable. La amplificación de las secuencias diana y de RNA-QS se realiza simultáneamente mediante un perfil universal de amplificación mediante PCR con unos pasos de temperatura y número de ciclos predefinidos. El reactivo de Master Mix incluye trifosfato de deoxiuridina (dUTP), en lugar de trifosfato desoxitimidina (dTTP), que se incorpora al ADN recién sintetizado (amplicón).20-22 La enzima AmpErase, que se incluye en la mezcla para PCR durante la primera ciclación térmica, elimina los amplicones contaminados de los procesos de PCR anteriores. Sin embargo, los amplicones nuevos no se eliminan porque la enzima AmpErase se inactiva cuando se expone a temperaturas superiores a los 55 °C.
El reactivo Master Mix de COBAS® HCV contiene sondas dobles de detección específicas para las secuencias diana del HCV y una para el RNA-QS. Las sondas están marcadas con marcadores emisores fluorescentes específicos para la diana que permiten la detección simultánea de fragmentos diana de HCV y RNA-QS en dos canales diana distintos.23, 24 Cuando no se une a la secuencia de la diana, la señal fluorescente de la sonda intacta se elimina mediante el marcador silenciador. Durante el paso de amplificación mediante PCR, la hibridación de las sondas con la plantilla específica de ADN mono-catenario provoca la escisión de la sonda por la actividad de la nucleasa 5’ a 3’ de la ADN polimerasa, lo que produce la separación de los marcadores emisor y silenciador, y la emisión de una señal fluorescente. Con cada ciclo de PCR, se generan cantidades crecientes de sondas escindidas y la señal acumulada del marcador emisor aumenta concomitante- mente. La detección y diferenciación en tiempo real de los productos de PCR se consigue mediante la cuantificación de la fluorescencia liberada por los marcadores emisores para las dianas víricas y el RNA-QS.